Mersin İlinde Kronik Hepatit B Hastalarında Hepatit B Virusu Genotiplerinin Belirlenmesi*
Determination of Hepatitis B Virus Genotypes in Chronic Hepatitis B Patients in Mersin Province, Turkey
Gürol
EMEKDAŞ1, Seda TEZCAN1, Gönül ASLAN1, Mehmet
Sami SERİN2, Orhan SEZGİN3,
Enver ÜÇBİLEK3, Mahmut ÜLGER1, Engin ALTINTAŞ3,
Aylin DÖĞEN2
1 Mersin Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Mersin.
1 Mersin University Faculty of Medicine, Department of Medical Microbiology, Mersin, Turkey.
2 Mersin Üniversitesi Eczacılık Fakültesi, Farmasötik Mikrobiyoloji Anabilim Dalı, Mersin.
2 Mersin University Faculty of Pharmacy, Department of Pharmaceutical Microbiology, Mersin, Turkey.
3 Mersin Üniversitesi Tıp Fakültesi, Gastroenteroloji Bilim Dalı, Mersin.
3 Mersin University Faculty of Medicine, Department of Gastroenterology, Mersin, Turkey.
* Bu çalışma Mersin Üniversitesi Bilimsel Araştırma Projeleri Birimi tarafından desteklenmiş olup [Proje No: BAP-SBE TM (ST) 2008-9 DR], Mersin Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalında Doktora tezi olarak hazırlanan çalışmanın bir bölümünü oluşturmaktadır. IV. Ulusal Viroloji Kongresi (23-26 Haziran 2011, İstanbul)'nde poster bildirisi olarak sunulmuştur.
ÖZET
Hepatit B virusu (HBV), nükleik asit dizi heterojenitesine göre tanımlanan on genotip (A-J) içermekte ve bu genotipler birbirlerinden oldukça farklı coğrafi dağılım göstermektedir. Ülkemizin değişik bölgelerinde yapılan çalışmalarda en yaygın olarak HBV genotip D saptanmaktadır; ancak bugüne kadar Mersin bölgesinde HBV genotipleri değerlendirilmemiştir. Bu çalışmada, Mersin ilindeki kronik hepatit B hastalarında, pre-S gen dizisi temel alınarak polimeraz zincir reaksiyonu-restriksiyon parça uzunluk polimorfizmi (PCR-RFLP) ve DNA dizi analizi ile HBV genotip dağılımının belirlenmesi amaçlanmıştır. Çalışmaya, Mersin Üniversitesi Tıp Fakültesi, Gastroenteroloji kliniğinde takip edilen 33'ü erkek, 21'i kadın ve yaş ortalamaları 40.05 yıl olan toplam 54 kronik hepatit B'li hastaya ait serum örnekleri alınmıştır. Hastalar, serum örneklerinde tespit edilebilir HBV DNA düzeyine sahip olup, en az bir yıldır antiviral tedavi almaktadır. HBV genotip tayini, HBV genomunun pre-S gen bölgesi PCR ile amplifiye edildikten sonra, AvaII ve MboI kesim enzimleri ile RFLP analizi uygulanarak gerçekleştirilmiştir. Doğrulama için, hasta örneklerinden 18'inin pre-S gen bölgesi PCR ürünlerine direkt DNA dizi analizi uygulanmıştır. Genotipler 43 NCBI (National Center for Biotechnology Information) referans HBV dizisi ile filogenetik olarak karşılaştırılarak belirlenmiştir. Genotip belirlenmesi 54 örneğin yedi tanesinde gerçekleştirilememiştir. Bu örneklerden üçü pre-S PCR ile negatif bulunmuş, üçü özgül olmayan bantlar vermiş, birinde ise PCR sonucunda beklenenden daha kısa, yaklaşık 300 bç hizasında delesyona uğramış bir PCR ürünü tespit edilmiştir. Kalan 47 örneğin PCR-RFLP analizinde dört farklı restriksiyon paterni belirlenmiştir. Bu paternlerden biri AvaII [-]/MboI [306/89/51] olup, örneklerin %72.3 (34/47)'ünde genotip D olarak açıkça ayrımı yapılmıştır. Diğer üç patern ise, AvaII [-]/MboI [357/306/89/51] (7/47, %14.9), AvaII [300/146]/MboI [306/89/51] (5/47, %10.7) ve AvaII [-]/MboI [357/89/---] (1/47, %2.1) olup, genotip ayrımı tam olarak yapılamamıştır. HBV dizilerinin filogenetik karşılaştırılması, bütün paternlere ait örneklerin NCBI genotip D dizileri içinde gruplandığını göstermiştir. AvaII [300/146]/MboI [306/89/51] ve AvaII [-]/MboI [357/89/---] paternleri ile delesyona uğramış örnek HBV izolatlarının pre-S gen varyantı olarak kabul edilmiştir. Bulgularımız, Mersin ilinde predominant HBV genotipinin, ülkemizde ve diğer Akdeniz ülkelerinde de yaygın olarak görülen tip olan, genotip D olduğunu göstermiştir. Çalışmamızın sonuçları ayrıca, ülkemizin güney bölgesinde dolaşımda olan HBV izolatlarının moleküler epidemiyolojisinin anlaşılmasında oldukça önemli olan, HBV izolatları arasındaki genotipik bir örnekliliği ve pre-S gen varyantlarının varlığını da ortaya koymuştur.
Anahtar sözcükler: Kronik hepatit B; hepatit B virusu; genotip; pre-S geni; RFLP; DNA dizi analizi.
ABSTRACT
Currently, ten genotypes (A-J) of hepatitis B virus (HBV) are identified based on the nucleic acid sequence heterogeneity, and these genotypes have been shown to have distinct geographic distribution. Reports of previous studies indicated that the genotype D is the predominant type among hepatitis B patients in different regions of Turkey, however there is no data for HBV genotypes to date from Mersin region. The aim of this study was to investigate the HBV genotypes by polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP) in chronic hepatitis B patients in Mersin province (located in the Mediterranean region of Turkey). A total of 54 serum samples were obtained from the chronic hepatitis B patients (33 male, 21 female; mean age: 40.05 years) followed-up at Gastroenterology Clinic of Mersin University Hospital. Patients had detectable HBV-DNA levels in their serum samples, and they were under antiviral therapy for at least one year. Genotyping of HBV was performed by RFLP analysis with the use of AvaII and MboI restriction enzymes after amplification of pre-S gene region by PCR. Confirmation of selected 18 cases was carried out with direct DNA sequencing. The genotypes were determined by phylogenetic comparison with 43 reference NCBI (National Center for Biotechnology Information) HBV sequences. Genotype determination was not successful in seven cases; since three of them were negative in preS-PCR, three of them yielded non-specific bands, and one of them exhibited a deleted PCR product, at the 300 bp level that was shorter than expected. Four different restriction patterns were determined in PCR-RFLP analysis of the remaining 47 samples. One of these patterns which was AvaII [-]/MboI [306/89/51], was clearly discriminated in 72.3% (34/47) of the samples as genotype D. Genotype discrimination of three patterns could not be done properly and these patterns were AvaII [-]/MboI [357/306/89/51] (7/47, 14.9%), AvaII [300/146]/MboI [306/89/51] (5/47, 10.7%), and AvaII [-]/MboI [357/89/---] (1/47, 2.1%). Phylogenetic comparison of HBV sequences demonstrated that all patterns in our cases were clustered in NCBI genotype D sequences. Patterns of AvaII [300/146]/MboI [306/89/51] and AvaII [-]/ MboI [357/89/---] and deleted sample were recognized as pre-S gene variants of HBV isolates. Our data indicated that the predominant HBV type was genotype D as commonly seen in Turkey and other Mediterranean countries. The results of this study also showed that the genotype uniformity and pre-S gene variants within the HBV isolates could be crucial in terms of understanding the molecular epidemiology of HBV circulating in the Mediterranean region of Turkey.
Key words: Chronic hepatitis B; hepatitis B virus; genotype; pre-S gene; RFLP; DNA sequencing.
Geliş Tarihi (Received): 30.11.2011 • Kabul Ediliş Tarihi (Accepted): 15.02.2012
GİRİŞ
Hepatit B virusu (HBV) enfeksiyonu dünya çapında bir sağlık sorunu olup, yaklaşık iki milyar kişinin HBV ile enfekte olduğu ve 350 milyon kişide enfeksiyonun kronikleştiği belirtilmektedir1. HBV enfeksiyonu, kendini sınırlayan akut hastalık ve inaktif taşıyıcılıktan, kronik hepatit, siroz ve hepatoselüler kansere kadar değişen geniş bir klinik spektruma sahiptir1.
Hepadnaviridae ailesinde yer alan HBV, zarflı ve ikozahedral simetrili bir DNA virusudur. Kısmi çift iplikli ve 3.2 kb büyüklüğündeki çembersel genom, birbirleriyle örtüşen dört gen bölgesi içerir ve virusun zarf (preS1, preS2, S), kor (C), polimeraz (P) ve X (X) proteinlerini kodlar2. HBV genom replikasyonu bir RNA aracısıyla olmakta ve DNA sentezi viral polimerazın ters (revers) transkriptaz (RT) aktivitesiyle gerçekleşmektedir. RT enziminin hata düzeltme (proof-reading) özelliği olmadığından, enfekte kişilerde oldukça geniş dizi çeşitliliğine sahip HBV popülasyonları ortaya çıkmaktadır3. Yapılan filogenetik analizlere göre bugün için HBV, tüm genomda %8'den daha fazla nükleotid dizi farklılığı dikkate alınarak A'dan J'ye kadar 10 farklı genotipe ayrılmıştır4,5,6. Bu genotiplerin dünyadaki dağılımı belirgin bir coğrafik ve etnik özellik göstermekte; genotipik farklılığının, prognoz ve tedaviye yanıt gibi hastalığın klinik özelliklerine katkıda bulunabileceği ileri sürülmektedir6,7,8. Örneğin; HBV genotip C veya D enfeksiyonunun, genotip A veya B'den daha yüksek replikasyon oranıyla siroz ve hepataselüler kanser gibi daha ciddi karaciğer hastalığı ile ilişkili olduğu belirtilmiştir6,9. Bu nedenle HBV genotiplerinin belirlenmesi, hastalığın ilerlemesi yönünden risk altında olan kronik hepatit B hastalarında daha iyi bir hasta yönetimi için oldukça önemlidir. HBV izolatlarının genotiplendirmesinde; restriksiyon parça uzunluk polimorfizmi (RFLP), DNA dizi analizi, filogenetik analiz, "line probe assay", ELISA ve polimeraz zincir reaksiyonu (PCR) temelli yöntemler kullanılabilmektedir7,8,9,10,11,12.
HBV enfeksiyonu açısından orta endemik bölgeler arasında yer alan Türkiye'de popülasyonun yaklaşık %5'i kronik hepatit B hastasıdır13,14. Ülkemizin değişik bölgelerinde yapılan çalışmalarda en yaygın olarak HBV genotip D saptanmaktadır15,16,17,18,19,20,21. Ancak şu ana kadar Mersin bölgesinde HBV genotipleri değerlendirilmemiştir. Bu çalışmada, Mersin ilinde HBV ile kronik olarak enfekte hastalarda, pre-S gen dizisi temel alınarak PCR-RFLP ve DNA dizi analizi ile HBV genotip dağılımının belirlenmesi amaçlanmıştır.
GEREÇ ve YÖNTEM
Hastalar
Çalışmaya, Mersin Üniversitesi Tıp Fakültesi, Gastroenteroloji kliniğinde takip edilen 33'ü erkek, 21'i kadın ve yaş ortalamaları 40.05 yıl olan toplam 54 kronik hepatit B'li hastaya ait serum örnekleri dahil edildi. Düzenli olarak kontrolü yapılan hastalar, en az bir yıldır antiviral tedavi almaktaydı. Çalışma, Mersin Üniversitesi Tıp Fakültesi Etik Kurul onayı ile gerçekleştirildi ve tüm hastaların bilgilendirilmiş onamı alındı. Hasta serumlarında HBV serolojik göstergeleri Architect i2000SR (Abbott Diagnostics, ABD), HBV DNA düzeyi ise Cobas TaqMan 48 (Roche Diagnostics, Almanya) sistemiyle tespit edilmişti.
HBV Genotiplerinin Belirlenmesi
Viral DNA izolasyonu: Serum örneklerinden viral DNA izolasyonu, modifiye klasik fenol-kloroform ve kloroform yöntemi kullanılarak gerçekleştirildi22. Öncelikle 150 µl serum örneği 450 µl parçalayıcı tampon [13.3 mmol/L Tris-HCl (pH 8.0), 6.7 µmol/µl etilen-diamin-tetra-asetik asit, %0.67 sodyum dodesil sülfat ve 133 mg/ml proteinaz-K] ile karıştırıldı ve 56°C'de bir gece boyunca inkübe edildi. İnkübasyonun sonunda önce iki kez fenol-kloroform (1:1) ekstraksiyonu, daha sonra bir kez kloroform ekstraksiyonu yapıldı. DNA, daha önceden soğutulmuş %96'lık etil alkol ile çöktürüldü. DNA pelleti havada kurutulduktan sonra 25 µl nükleaz içermeyen steril suda çözdürüldü ve kalıp DNA olarak kullanılmak üzere -20°C'de saklandı.
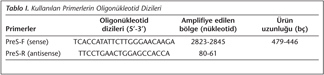
PCR-RFLP analizi: HBV genotiplerinin belirlenmesinde, Lindh ve arkadaşlarının23 tarif ettiği PCR-RFLP yöntemi kullanıldı25. HBV genomunun pre-S gen bölgesinin 2823. nükleotid ve 80. nükleotid arasında kalan 479 baz çift (bç)'lik segmenti, özgül primerler kullanılarak PCR ile amplifiye edildi (Tablo I). HBV genotiplerinin belirlenmesi, amplifiye segmentin AvaII (5'...G6GWCC...3', W= A veya T) ve MboI (5' 6GATC...3') kesim enzimleriyle inkübe edilerek RFLP analizinin uygulanmasıyla gerçekleştirildi.
Her bir örneğin PCR amplifikasyonu 50 µl'lik reaksiyon hacimlerinde (5 µl 10XPCR tampon, 2.5 µmol/µl MgCl2, 0.2 µmol/µl dNTP karışımı, 0.25 pmol/µl her bir primer, 1.25 U Taq DNA polimeraz, 5 µl örnek DNA'sı) gerçekleştirildi. Isı döngü cihazında (Eppendorf, Mastercycler, Almanya) amplifikasyon koşulları; 94°C'de 10 dakika başlangıç denatürasyonu, 40 döngü 94°C'de 45 saniye denatürasyon, 60°C'de 1 dakika bağlanma, 72°C'de 1.5 dakika uzama ve 70°C'de 7 dakika son uzama şeklinde uygulandı. PCR ürünleri, %1'lik agaroz jel elektroforezinden sonra 0.5 µg/ml etidyum bromür ile boyandıktan sonra ultraviyole transilüminatörde görüntülendi.
RFLP analizi için, ayrı reaksiyon tüplerinde, pozitif PCR ürünlerinden 8 µl'si 2 ünite (0.2 µl) AvaII (Fermentas, #ER0311) ve MboI (Fermentas, #ER0812), 1 µl 10X kesim tamponu (Buffer R) ve 0.8 µl nükleaz içermeyen steril su ile karıştırıldıktan sonra 37°C'de 4-16 saat inkübe edildi. Kesim ürünleri agaroz jel elektroforezi sonrasında ultraviyole transilüminatörde görüntülendi. Kesilen bantların büyüklüğü moleküler ağırlık standardı (GeneRuler 100 bç DNA ladder, #SM0241, Fermentas) ile kıyaslanarak değerlendirildi.
PCR ürünlerinin direkt DNA dizi analizi: Restriksiyon paternlerinin değerlendirilmesi ve beklenmedik paternlerin restriksiyon alanının doğrulanması için amplifiye pre-S gen bölgesi ürünlerine direkt DNA dizi analizi uygulandı. Toplam 18 örneğe ait pre-S gen bölgesi ürünlerinin "sense" ve "antisense" dizileri "Bigdye Terminator v3.1Cycle Sequencing kit" (Applied Biosystems, ABD) kullanılarak dizi analizi gerçekleştirildi. Dizi analizi verileri ABI PRISM 3130XL Genetic Analyzer (Applied Biosystems, ABD) kromatogram şeklinde toplandı.
Her örneğe ait dizi analizi verileri CLUSTAL X versiyon 1.83 yazılım programı ile hizalandıktan sonra GENDOC versiyon 2.6.002 (Multiple Sequence Alignment Editor & Shading Utility) yazılım programı ile konsensus dizi olarak kaydedildi.
Filogenetik analiz: Hasta örneklerinden 18 tanesi ve 43 referans NCBI (National Center for Biotechnology Information) dizisi (29'u genotip D, 14'ü diğer genotipler) olmak üzere toplam 61 dizinin filogenetik karşılaştırması MEGA versiyon 3.1 paket programı kullanılarak gerçekleştirildi. Dizi analizinden elde edilen pre-S gen bölgesi dizisi CLUSTAL X programında, referans diziler ile toplu bir şekilde hizalandıktan sonra, GENEDOC programında gerekli düzeltmeler yapıldı. Filogenetik ağaç MEGA versiyon 3.1 yazılım programı kullanılarak "Neighbor-Joining" metodu ile oluşturuldu.
Restriksiyon alanlarının doğrulanması için Vector NTI 9.0 (InforMax, Frederick, MD) yazılım programında dizi analizleri yapılan örneklerin herbirinin ve GenBank dizilerinin AvaII ve MboI restriksiyon alanlarının haritası çıkarıldı ve görünen paternleri oluşturan fragment büyüklükleri hesaplandı.
BULGULAR
Çalışmaya dahil edilen 54 hastanın tümü HBsAg ve anti-HBc IgG pozitif olup; 19 (%35.2)'unda HBeAg, 4 (%7.4)'ünde anti-HBc IgM ve 2 (%3.7)'sinde anti-HBs pozitif bulunmuştur. HBV DNA düzeyi ise HBeAg pozitif hastalarda 15300-789000 (ortanca: 52500) IU/ml iken, HBeAg negatiflerde 84.7-23950 (ortanca: 2350) IU/ml arasında saptanmış, bir hastada HBV DNA kantite edilememiştir.
PCR-RFLP Sonuçları
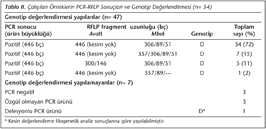
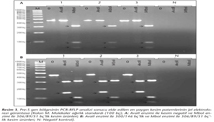
Çalışmamızda, pre-S PCR yapılan bütün örnekler yaklaşık 446 bç uzunluğunda ürün vermiş ve bu örneklerin genotip D olduğu düşünülmüştür. RFLP ile HBV genotip belirlenmesi 54 örneğin 47'sinde yapılmış; üç örneğin pre-S PCR sonucu negatif olduğundan, üç örnekte özgül olmayan bantlar gözlendiğinden ve bir örnekte beklenenden daha kısa, yaklaşık 300 bç hizasında delesyonlu ürün saptandığından toplam yedi örnekte genotipleme yapılamamıştır (Tablo II) (Resim 1). PCR'si negatif bulunan üç hastanın ikisinde HBV DNA düzeyi < 6, birinde 9180 IU/ml olarak belirlenmiş; PCR'de özgül olmayan ürünlerin gözlendiği üç hastada HBV DNA düzeyi, 5.170.000, 229.000 ve 22.600 IU/ml olarak izlenmiştir. PCR'de delesyonlu amplifikasyon ürünü veren hastanın viral yükü ise 27.300.000 IU/ml'dir.
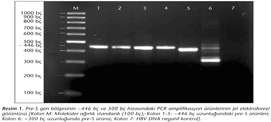
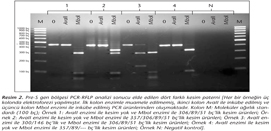
Genotip ayrımı, pre-S PCR ürünlerinin AvaII ve MboI enzimleriyle kesimini takiben %1.5'lik agaroz jel elektroforezinde oluşan fragmentlerin sayı ve uzunluğunun değerlendirilmesiyle gerçekleştirilmiş ve dört farklı restriksiyon paterni belirlenmiştir (Resim 2). Bu paternlerden biri AvaII [-]/MboI [306/89/51] olup, örneklerin %72.3 (34/47)'ünde genotip D olarak açıkça ayrımı yapılmıştır. Diğer üç patern AvaII [-]/MboI [357/306/89/51] (7/47, %14.9), AvaII [300/146]/MboI [306/89/51] (5/47, %10.7) ve AvaII [-]/MboI [357/89/---] (1/47, %2.1) olup, beklenmeyen bu kesim paternleriyle genotip ayrımı kesin olarak yapılamamıştır. En yaygın görülen kesim paternlerinden AvaII [-]/MboI [306/89/51] ile AvaII [300/146]/MboI [306/89/51] Resim 3'te gösterilmiştir.
Filogenetik Analiz Sonuçları
Beklenmeyen paternlerin genotip değerlendirmesinin kesin olarak yapılabilmesi için, bu beklenmeyen patern veren örneklere (13 adet), delesyona uğramış yaklaşık 300 bç'lik örneğe ve RFLP ile genotip D olarak belirlenebilenlerin doğrulanması için temsilen seçilen dört örnek olmak üzere toplam 18 örneğe DNA dizi analizi uygulanmıştır. Çalışılan 18 hasta ve 43 referans NCBI HBV dizisinin (29'u genotip D ve 14'ü diğer genotipler) filogenetik karşılaştırması, bütün paternlere ait HBV dizilerinin, NCBI genotip D dizileri içinde gruplandığını göstermiştir (Şekil 1).
AvaII enzimi ile kesilmeyen ve MboI enzimi ile 306/89/51 bç'lik kesim ürünleri veren 34 örneği temsilen, bu örneklerden seçilen dört HBV pre-S gen bölgesi, NCBI genotip D dizilerindeki gibi AvaII için restriksiyon alanına sahip olmadığı ve MboI için pre-S geninin 2943. ve 28. pozisyonlarında restriksiyon alanına sahip olduğu belirlenmiştir. AvaII enzimiyle kesilmeyen ve MboI enzimiyle 357/306/89/51 bç'lik kesim ürünleri veren yedi örnekte de, genotip D olarak belirlenenlerde olduğu gibi pre-S geninin 2943. ve 28. pozisyonlarında restriksiyon alanına sahip olduğu gösterilmiştir. Bu örneklerde gözlenen 357 bç'lik fragmentin, 306 ve 51 bç'lik fragmentleri oluşturan kesilmemiş bir fragment olduğu anlaşılmıştır. AvaII enzimiyle 300/146 bç ve MboI enzimiyle 306/89/51 bç'lik kesim ürünleri veren beş örneğin restriksiyon haritasında bu kesim alanları doğrulanmıştır. Restriksiyon haritası çıkarılan NCBI dizilerinden HBV genotip D2 olarak belirlenen Türkiye'ye ait AY796031 referans dizisinde ve genotip D1 olarak belirlenen İran'a ait AY741796 referans HBV dizisinde de bu patern gösterilmiştir. Bu sebeple bu beş HBV dizisi genotip D olarak belirlenmiştir. AvaII enzimiyle kesilmeyen ve MboI enzimiyle 357/89/-- bç'lik kesim ürünleri veren bir örneğin dizi analizi sonrasında diğer gen dizileriyle hizalandığında MboI restriksiyon alanlarından birinin kaybolmasına neden olan 31. nükleotiddeki G'den A'ya nükleotid değişimi meydana geldiği görülmüştür.
Delesyona uğramış ~300 bç'lik ürün veren örneğin DNA dizi analizinde, bütün genotip D olarak belirlenen HBV pre-S dizilerinde bulunan 33 bç'lik delesyon görülmüş; bu nedenle genotip D olarak değerlendirilmiştir. Ayrıca filogenetik analizinde genotip D grubu içinde yer aldığı gözlenmiştir. Ayrıca bu örnekte delesyona uğrayan bölgenin, pre-S gen bölgesinin 3015-3026, 3030-3041 ve 3122-80. nükleotidler arasında olduğu belirlenmiştir. Çalışılan tüm hastaların HBV genotiplerinin D olduğu görülmüştür (Tablo II).
TARTIŞMA
HBV genotipleri, tüm dünyada değişik coğrafik dağılım paterni gösterir3,7,8. Bunlardan genotip A, genelde Avrupa, Kuzey Amerika, Brezilya ve Güney Afrika'da; genotip B ve C benzer bir dağılım ile Güney-Doğu Asya, Uzak Doğu ve Pasifik bölgelerinde; genotip D, Güney Avrupa, Orta Asya, Hindistan ve Akdeniz bölgesinde; genotip E, Batı ve Güney Afrika'da; genotip F, Güney ve Orta Amerika'da; genotip G, Amerika ve Avrupa (Fransa)'da; genotip H, Meksika ve Kaliforniya'da; genotip I ise Çin ve Vietnam'da yaygın olarak bulunmaktadır24,25,26,27,28,29,30. Uluslararası seyahat ve insan göçlerinin oldukça sık olduğu günümüzde, yeni HBV genotiplerinin toplumlara girişi, genotipler arası rekombinasyona veya bir genotipin diğerine değişmesine neden olabilmektedir31,32. Göçler veya çeşitli genotiplerin kıtalar arası dolaşımı göz önüne alındığında, HBV genotiplerinin belirlenmesi, Avrupa ve Asya kıtaları arasında bir köprü olan ülkemiz için büyük öneme sahiptir. Ayrıca son yıllarda, HBV genotipleri ile hastalığın şiddeti, prognozu ve tedaviye yanıtı arasında ilişki olduğu vurgulanmakta; genotip A'nın kronik enfeksiyona ilerlemede, genotip C'nin ise siroz ve HSK gibi ciddi hastalıkların oluşmasında diğer genotiplerden daha yüksek risk taşıdığı ifade edilmektedir8,33,34. Ancak çalışmamızda, Mersin ilinde kronik HBV enfeksiyonlu 54 hastanın hepsinin genotip D ile enfekte olduğu belirlendiğinden, kronik enfeksiyon ile genotip arasındaki ilişki değerlendirilememiştir.
HBV genotiplerinin genom büyüklüğü ve nükleotid pozisyonları da farklılık göstermektedir. Genotip B, C, F ve H, 3215 nükleotid uzunluğunda iken; genotip D pre-S1 gen bölgesinde 33 nükleotidlik, genotip E ve G de aynı bölgede 3 nükleotidlik delesyona sahiptir. Buna karşın genotip A, polimeraz geninin terminal protein bölgesinde 6 nükleotidlik, genotip G ise kor geninin amino ucunda 36 nükleotidlik insersiyona sahiptir7. Lindh ve arkadaşları23, bütün NCBI genotip D suşlarının pre-S1 gen bölgesinde 2859-2891. nükleotid pozisyonunda sahip olduğu 33 bç'lik bu delesyon nedeniyle, PCR sonrası bu suşların 479 bç yerine 446 bç uzunluğunda ürün verdiğini ve böylece genotip D suşlarının tespit edilmesinin kolaylaştığını ifade etmektedirler. Bizim çalışmamızda da HBV genotiplerinin belirlenmesinde Lindh ve arkadaşlarının23 tanımladığı PCR-RFLP temelli yöntem kullanılmıştır. Yaptığımız çalışmada genotiplendirmesi yapılabilen hastaların hepsi yaklaşık 446 bç uzunluğunda (33 bç delesyonlu) ürün vermiş; ancak bir örnek yaklaşık 300 bç uzunluğunda delesyonlu ürün vermiştir. Bu örnekte 446 bç hizasında da PCR ürünü görünmekte olup, dolaşımda "wild" tip ve delesyona uğramış varyantlardan oluşan karışık virus havuzunun olduğunu düşündürmektedir. Bu metotta, bütün genotip D dizileri, AvaII enzimi için restriksiyon alanı bulunmadığı için bu enzimle kesilmemekte; MboI enzimiyle kesimden sonra ise iki temel restriksiyon paterni (306/67/52/21 ve 306/88/52) elde edilmektedir23. Ancak, yaptığımız bu çalışmada ilginç bir şekilde, dört farklı restriksiyon paterni belirlenmiştir. Bu paternlerden üçü beklenmedik restriksiyon paternleri olup, çalışılan örneklerin 13 tanesinde (tüm örneklerin %27.7'si) görülmüştür. Bu beklenmedik restriksiyon paternleri ise orijinal makalede bildirilmemiştir (Tablo II). Genotip D olarak ayrımı yapılabilen patern AvaII [-]/MboI [306/89/51] olup örneklerin %72.3 (34/47)'ünde tespit edilmiştir. Diğer üç restriksiyon paterni olan AvaII [-]/MboI [357/306/89/51] (7/47, %14.9), AvaII [300/146]/MboI [306/89/51] (5/47, %10.7) ve AvaII [-]/MboI [357/89/---] (1/47, %2.1)'nin RFLP ile genotip ayrımı kesin olarak gerçekleştirilememiştir. Beklenmeyen restriksiyon paterni veren 13 örneğin, bir delesyonlu örneğin ve PCR-RFLP ile ayrımı yapılabilen örneklerden kontrol olarak temsilen seçilen dört örneğin ve 43 referans HBV dizisinin filogenetik analizi, bütün paternlere ait örneklerin genotip D dizileri arasında gruplandığını göstermiştir.
AvaII [-]/MboI [357/306/89/51] paterni veren pre-S gen dizilerinin restriksiyon alanı analizi sonunda, 357 bç'lik fragmentin 306 ve 51 bç'lik fragmentleri oluşturan kesilmemiş PCR fragmenti olduğu anlaşılmıştır. Her ne kadar 306 ve 51 bç'yi oluşturan restriksiyon alanları doğrulanmış olsa da, bu örnekler MboI ile kesilebilen ve kesilemeyen karışık virus havuzunu (türümsü) içerebilir. Karışık izolatların bulunduğu bu örneklerde ise, ancak klonlama yaparak gerçek virus popülasyonları gösterilebilir. AvaII [300/146]/MboI [306/89/51] paterni veren beş örneğin, bu restriksiyon alanları doğrulanmıştır. Bu paterni oluşturan restriksiyon alanları, Türkiye AY796031 genotip D2 ve İran AY741796 genotip D1 referans dizilerinde de gösterilmiştir. Bu sebeple bu beş örnek genotip D olarak kesin olarak belirlenmiştir. AvaII [-]/MboI [357/89/---] paterninin analizinde, 28. nükleotid pozisyonunda MboI'in kesim alanının değişmesine sebep olan tek nükleotid polimorfizmi gözlenmiştir. Görüldüğü gibi, RFLP yönteminin en önemli dezavantajlarından biri; özgül restriksiyon alanının değişmesine sebep olan tek nükleotid değişimleridir. Özellikle bu nükleotid değişiklikleri özgün restriksiyon alanlarında meydana geldiğinde, HBV suşlarının genotip tayini başarısız olmakta ve bu da RFLP'nin genotiplerin belirlenmesindeki güvenilirliğini azaltmaktadır.
Çalışmamızda, tüm hastalardaki HBV suşlarının genotip D olduğu görülmüş; pre-S gen varyantlarının varlığı filogenetik analizle gösterilmiştir. Bu suşların prevalansı Mersin ili için %14.6 (7/48) olarak bulunmuştur; ancak bunun diğer coğrafi bölgeler için de belirlenmesi gerekmektedir. Çalışmamızın sonuçları, genotip D dizilerinde pre-S gen bölgesinde AvaII enzimiyle kesilebilen, 300/146 bç paternine karşılık gelen ve daha önceden bildirilmemiş yeni restriksiyon alanını göstermiştir. AvaII [300/146]/MboI [306/89/51] ve AvaII [-]/MboI [357/89/---] paternleri ile (pre-S gen bölgesinin 3015-3026, 3030-3041 ve 3122-80. nükleotidler arasında) delesyona uğramış örnek, HBV genotip D suşlarının pre-S gen varyantı olarak kabul edilmiştir. Utama ve arkadaşlarının35 çalışmasında, Endonezya'da delesyonlu izolatlara %12.1 oranında rastlandığı ve belirlenen pre-S gen delesyonlu suşların hastalığın ilerlemesiyle ilişkili olduğu belirtilmiştir. Bu sebeple, çalışmada elde ettiğimiz verilerin, bölgemizde hastalığın tedavisi başta olmak üzere, HBV'nin immünolojik ve genetik tanısına ışık tutacağı düşüncesindeyiz.
Ülkemizin çeşitli bölgelerinde, pre-S veya S gen bölgesinin PCR-RFLP veya DNA dizi analizi ile yapılan HBV genotiplendirme çalışmalarında en sık rastlanılan genotip D'dir15,16,17,18,19,20,21,36,37,38,39,40. Serin ve arkadaşlarının17 çalışmasında, Türkiye'nin güney bölgesinde HBV genotiplerinin genel dağılımı araştırılmış, ancak bu çalışmada Mersin ili yer almamıştır. Yaptığımız bu çalışmada, ilimizde ilk defa HBV izolatlarının genotipleri tespit edilerek pre-S gen varyantları gösterilmiş; çalışılan 47 kronik HBV enfeksiyonlu hastada en yaygın genotipin, ülkemiz ve bölgemiz verileriyle uyumlu olarak genotip D olduğu belirlenmiştir. Buna karşın ülkemizde diğer HBV genotiplerinin bildirildiği çalışmalar da vardır. Onganer ve arkadaşları41, İstanbul'da HBV ile enfekte 50 hastada, S gen bölgesinin RFLP analizi ile genotip D (%84), A (%12) ve F (%4) olmak üzere üç farklı genotip saptamışlardır. Kaya ve arkadaşları18 ise Isparta'da 124 hastada yaptıkları çalışmada, pre-S2 epitop ELISA yöntemiyle genotip D/E (%85.1), A (%4.4), C (%1.4) ve F (%0.7)'yi tespit etmişlerdir. Bu sonuçlar Türkiye'de dolaşımda olan HBV suşlarının heterojenitesini göstermektedir. Ancak ilginç olarak, pre-S gen bölgesinin hedef alındığı DNA dizi analizi çalışmalarda sadece D genotipi bildirilirken, S geninin PCR-RFLP analizi veya pre-S2 epitop ELISA'nın kullanıldığı çalışmalarda genotip A, C ve F gibi farklı genotiplerin bulunduğu görülmektedir. Dolayısıyla, DNA dizi analizi ile desteklenmeyen yöntemlerde, varyant suşlar veya şüpheli sonuçların farklı değerlendirmelere sebep olabileceğinin göz önünde bulundurulması gerekmektedir.
Ülkemizde kronik hepatit B'nin tedavisinde HBV genotiplerinin belirlenmesinin, tedaviye başlama veya hasta yönetiminde kullanılmasıyla ilgili bir kriter bulunmamaktadır. HBV genotip dağılımıyla ilgili ülkemizde yapılan çalışmalarda homojen olarak sadece D genotipinin gösterilmiş olması, standart klinik yaklaşım kolaylığı sağlayabilmiştir. Ayrıca hakim olarak sadece bir genotipin görülmesi de farklı genotipler ile koenfeksiyon olasılığını ve genotipler arası olası rekombinasyon riskini de sınırlamaktadır. Çalışmada elde ettiğimiz genotip bir örnekliliği ile ilgili moleküler epidemiyolojik verilerin kronik hepatit B'li hastalara karşı klinik yaklaşıma katkıda bulunacağı düşünülmektedir.
Sonuç olarak çalışmamızda, Türkiye'nin birçok bölgesinde ve diğer Akdeniz ülkelerinde olduğu gibi, kronik hepatit B hastalarında en yaygın HBV genotipinin D olduğu gösterilmiş ve RFLP yönteminin kolay uygulanabilir ve görece olarak başarılı olması (örneklerin %72.3'ünde genotip ayrımı kesin olarak yapılmıştır) nedeniyle klinik tanısal laboratuvarlarda kullanılabileceği kanısına varılmıştır. DNA dizi analizi ve filogenetik analiz ile nükleotid dizilerinin çıkarılarak HBV genotiplerinin belirlenmesi, oldukça güvenilir sonuçlar verse de, bizim gibi sınırlı kaynaklara sahip moleküler mikrobiyoloji laboratuvarlarında, rutin tanısal test olarak kullanılması uygun değildir. Diğer yandan, pre-S gen varyantlarının tespit edilebilmesi için DNA dizi analizi ve filogenetik analizin, ülkemizde dolaşımda olan HBV suşlarının moleküler epidemiyolojisinin anlaşılmasında uygun yöntemler olduğu düşünülmüştür.
KAYNAKLAR
- Nebbia G, Peppa D, Maini MK. Hepatitis B infection: current concepts and future challenges. QJM 2012; 105(2): 109-13. [Özet]
- Kreutz C. Molecular, immunological and clinical properties of mutated hepatitis B viruses. J Cell Mol Med 2002; 6(1): 113-43. [Özet]
- Echevarría JM, Avellón A. Hepatitis B virus genetic diversity. J Med Virol 2006; 78(Suppl 1): S36-42. [Özet]
- Cao GW. Clinical relevance and public health significance of hepatitis B virus genomic variations. World J Gastroenterol 2009; 15(46): 5761-9. [Özet] [Tam Metin] [PDF]
- Yu H, Yuan Q, Ge SX, et al. Molecular and phylogenetic analyses suggest an additional hepatitis B virus genotype "I". PLoS One 2010 19; 5(2): e9297. [Özet] [Tam Metin] [PDF]
-
Kao JH. Molecular epidemiology of hepatitis B virus. Korean J Intern
Med 2011; 26(3): 255-61.
[Özet] [Tam Metin] [PDF] - Bartholomeusz A, Schaefer S. Hepatitis B virus genotypes: comparison of genotyping methods. Rev Med Virol 2004; 14(1): 3-16. [Özet]
- Lin CL, Kao JH. The clinical implications of hepatitis B virus genotype: recent advances. J Gastroenterol Hepatol 2011; 26(Suppl 1): 123-30. [Özet] [Tam Metin] [PDF]
- Chu CJ, Lok AS. Clinical significance of hepatitis B virus genotypes. Hepatology 2002; 35(5): 1274-76.
- Valsamakis A. Molecular testing in the diagnosis and management of chronic hepatitis B. Clin Microbiol Rev 2007; 20(3): 426-39. [Özet] [Tam Metin] [PDF]
- Liu WC, Lindh M, Buti M, et al. Genotyping of hepatitis B virus genotypes A to G by multiplex polymerase chain reaction. Intervirology 2008; 51(4): 247-52. [Özet]
- Ali MM, Hasan F, Ahmad S, Al-Nakib W. Comparative evaluation of INNO-LiPA HBV assay, direct DNA sequencing and subtractive PCR-RFLP for genotyping of clinical HBV isolates. Virol J 2010; 7: 111. [Özet] [Tam Metin] [PDF]
- Uzunalimoglu O, Yurdaydin C, Cetinkaya H, et al. Risk factors for hepatocellular carcinoma in Turkey. Dig Dis Sci 2001; 46(5): 1022-8. [Özet]
- Mıstık R, Balık I. Türkiye'de viral hepatitlerin epidemiyolojik analizi, s: 9-55. Tekeli E, Balık I (ed), Viral Hepatit 2003. Hepatitle Savaşım Derneği, İstanbul.
- Leblebicioglu H, Eroglu C; Members of the Hepatitis Study Group. Acute hepatitis B virus infection in Turkey: epidemiology and genotype distribution. Clin Microbiol Infect 2004; 10(6): 537-41. [Özet] [Tam Metin] [PDF]
- Bozdayi G, Türkyilmaz AR, Idilman R, et al. Complete genome sequence and phylogenetic analysis of hepatitis B virus isolated from Turkish patients with chronic HBV infection. J Med Virol 2005; 76(4): 476-81. [Özet]
- Serin MS, Akkiz H, Abayli B, Oksuz M, Aslan G, Emekdas G. Genotyping of hepatitis B virus isolated from chronic hepatitis B patients in the South of Turkey by DNA cycle-sequencing method. Diagn Microbiol Infect Dis 2005; 53(1): 57-60. [Özet]
- Kaya S, Cetin ES, Aridogan BC, Onal S, Demirci M. Distribution of hepatitis B virus (HBV) genotypes among HBV carriers in Isparta. Iran Biomed J 2007; 11(1): 59-63. [Özet]
- Sertoz RY, Erensoy S, Pas S, Ozacar T, Niesters H. Restriction fragment length polymorphism analysis and direct sequencing for determination of HBV genotypes in a Turkish population. New Microbiol 2008; 31(2): 189-94. [Özet]
- Külah C, Cirak MY. Determination of hepatitis B virus genotypes by DNA sequence analysis in patients from Ankara, Turkey. Mikrobiyol Bul 2010; 44(2): 245-53. [Özet] [PDF]
- Atalay MA, Gokahmetoglu S, Aygen B. Genotypes of hepatitis B virus in Central Anatolia, Kayseri, Turkey. Saudi Med J 2011; 32(4): 360-3. [Özet]
- Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. Book 3, 1989, 2nd ed. Cold Spring Harbor Laboratory Press, New York.
- Lindh M, Gonzalez JE, Norkrans G, Horal P. Genotyping of hepatitis B virus by restriction pattern analysis of a pre-S amplicon. J Virol Methods 1998; 72(2): 163-74. [Özet]
- Norder H, Hammas B, Lee SD, et al. Genetic relatedness of hepatitis B viral strains of diverse geographical origin and natural variations in the primary structure of the surface antigen. J Gen Virol 1993; 74(Pt 7): 1341-8. [Özet] [PDF]
- Stuyver L, De Gendt S, Van Geyt C, et al. A new genotype of hepatitis B virus: complete genome and phylogenetic relatedness. J Gen Virol 2000; 81(Pt 1): 67-74. [Özet] [Tam Metin] [PDF]
- Arauz-Ruiz P, Norder H, Robertson BH, Magnius LO. Genotype H: a new Amerindian genotype of hepatitis B virus revealed in Central America. J Gen Virol 2002; 83(Pt 8): 2059-73. [Özet] [Tam Metin] [PDF]
- Chang M. Hepatitis B virus mutation in children. Indian J Pediatr 2006; 73(9): 803-7. [Özet]
- Alvarado-Esquivel C, Sablon E, Conde-González CJ, et al. Molecular analysis of hepatitis B virus isolates in Mexico: predominant circulation of hepatitis B virus genotype H. World J Gastroenterol 2006; 12(40): 6540-5. [Özet]
- Ayed K, Gorgi Y, Ayed-Jendoubi S, et al. Hepatitis B virus genotypes and precore/core-promoter mutations in Tunisian patients with chronic hepatitis B virus infection. J Infect 2007; 54(3): 291-7. [Özet]
- Victoria Fda S, Oliveira CM, Victoria MB, Victoria CB, Ferreira LC. Characterization of HBeAg-negative chronic hepatitis B in western Brazilian Amazonia. Braz J Infect Dis 2008; 12(1): 27-37. [Özet]
- Banerjee A, Datta S, Chandra PK, Roychowdhury S, Panda CK, Chakravarty R. Distribution of hepatitis B virus genotypes: phylogenetic analysis and virological characteristics of genotype C circulating among HBV carriers in Kolkata, Eastern India. World J Gastroenterol 2006; 12(37): 5964-71. [Özet]
- Scotto G, Martinelli D, Di Tullio R, Fazio V. Epidemiological and clinical features of hepatitis B virus genotypes among immigrants in Southern Italy. Hepat Res Treat 2010; 2010: 878356. [Özet] [Tam Metin] [PDF]
- Suzuki Y, Kobayashi M, Ikeda K, et al. Persistence of acute infection with hepatitis B virus genotype A and treatment in Japan. J Med Virol 2005; 76(1): 33-9. [Özet]
- Yang HI, Yeh SH, Chen PJ, et al. Associations between hepatitis B virus genotype and mutants and the risk of hepatocellular carcinoma. J Natl Cancer Inst 2008 20; 100(16): 1134-43. [Özet] [Tam Metin] [PDF]
- Utama A, Siburian MD, Fanany I, et al. Low prevalence of hepatitis B virus pre-S deletion mutation in Indonesia. Med Virol 2011; 83(10): 1717-26. [Özet]
- Yalcin K, Degertekin H, Bahcecioglu IH, et al. Hepatitis B virus genotype D prevails in patients with persistently elevated or normal ALT levels in Turkey. Infection 2004; 32(1): 24-9. [Özet]
- Ozdemir FT, Duman D, Ertem D, et al. Determination of hepatitis B genotypes in patients with chronic hepatitis B virus infection in Turkey. Turk J Gastroenterol 2005; 16(4): 183-7. [Özet]
- Sunbul M, Leblebicioglu H. Distribution of hepatitis B virus genotypes in patients with chronic hepatitis B in Turkey. World J Gastroenterol 2005; 11(13): 1976-80. [Özet]
- Aksoy A, Ozdarendeli A. Genotyping of hepatitis B virus by restriction enzyme analysis. Mikrobiyol Bul 2006; 40(3): 215-23. [Özet] [PDF]
- Kalayci R, Altindiş M, Gülamber G, Demirtürk N, Akcan Y, Demirdal T. Genotype distribution of chronic hepatitis B and hepatitis C patients and investigation of the resistance patterns in hepatitis B cases. Mikrobiyol Bul 2010; 44(2): 237-43. [Özet] [PDF]
- Onganer PU, Oguzoglu N, Ozer A. Mutation and genotype analysis of hepatitis B virus on acute and chronic infected selected patients in Turkey. J Cell Mol Biol 2006; 5(1): 33-42.
İletişim (Correspondence):
Yrd. Doç. Dr. Seda Tezcan,
Mersin Üniversitesi Tıp Fakültesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
Yenişehir, 33169 Mersin, Türkiye.
Tel (Phone): +90 324 341 2815-1576,
E-posta (E-mail): tezcanseda@yahoo.com