Türkiye'de HIV-1 ile Enfekte Olgularda APOBEC3 Hipermutasyonları
APOBEC3 Hypermutations in HIV-1 Infected Cases in Turkey
Murat SAYAN1, Funda ŞİMŞEK2, Nurgül CERAN3, Başak DOKUZOĞUZ4, Haluk ERAKSOY5
1 Kocaeli Üniversitesi Tıp Fakültesi Hastanesi, Merkez Laboratuvarı, PCR Ünitesi, Kocaeli.
1 Kocaeli University Medical Faculty Hospital, Central Laboratory, PCR Unit, Kocaeli, Turkey.
2 İstanbul Okmeydanı Eğitim ve Araştırma Hastanesi, Enfeksiyon Hastalıkları ve Klinik Mikrobiyoloji Kliniği, İstanbul.
2 Istanbul Okmeydani Training and Research Hospital, Infectious Diseases and Clinical Microbiology Clinic, Istanbul, Turkey.
3 Haydarpaşa Numune Eğitim ve Araştırma Hastanesi, Enfeksiyon Hastalıkları ve Klinik Mikrobiyoloji Kliniği, İstanbul.
3 Haydarpasa Training and Research Hospital, Infectious Diseases and Clinical Microbiology Clinic, Istanbul, Turkey.
4 Ankara Numune Eğitim ve Araştırma Hastanesi, Enfeksiyon Hastalıkları ve Klinik Mikrobiyoloji Kliniği, Ankara.
4 Ankara Numune Training and Research Hospital, Infectious Diseases and Clinical Microbiology Clinic, Ankara, Turkey.
5 İstanbul Üniversitesi İstanbul Tıp Fakültesi, Enfeksiyon Hastalıkları ve Klinik Mikrobiyoloji Anabilim Dalı, İstanbul.
5 Istanbul University Istanbul Faculty of Medicine, Department of Infectious Diseases and Clinical Microbiology,
Istanbul, Turkey.
ÖZET
Konak genetik faktörleri insan immün yetmezlik virusu (HIV)'nun patogenezi üzerinde etkili olabilmektedir. Bunlardan biri olan APOBEC3 (apolipoprotein B mRNA editing enzyme catalytic polypeptide-like 3) proteinleri, HIV vif (virion infektivite faktörü) aksesuvar proteini ile sürekli bir etkileşim içinde olup, HIV ters transkripsiyonunda oluşturduğu hasar ile antiretroviral aktivite göstermektedir. Bu çalışmada, ülkemizde elde edilen HIV-1 izolatlarında APOBEC 3G/F hipermutasyonlarının tanımlanması amaçlanmıştır. Çalışma grubu, Haziran 2009-Şubat 2012 tarihleri arasında, yeni tanı almış 394 hasta [349 erkek, 45 kadın; ortanca yaş (aralık): 37.1 (2-69) yıl; ortanca CD4+ T-hücre sayısı (aralık): 340 (1-1660) mm3; ortanca HIV-RNA yükü (aralık): 5.76 + E5 (8.7 + E2-9.4 + E6) IU/ml] ile HAART tedavisi altında 121 hasta [99 erkek, 22 kadın; ortanca yaş (aralık): 40.7 (20-70) yıl; ortanca CD4+ T-hücre sayısı (aralık): 195 (6-720) mm3; ortanca HIV-RNA yükü (aralık): 5.4 + E5 (1.37 + E3-1.07 + E7) IU/ml] olmak üzere toplam 515 HIV-1 pozitif hastadan oluşturulmuştur. HIV-1 pol geninin ters transkriptaz (kodon 41-238) ve proteaz kangalları (kodon 1-99) nested RT-PCR ile çoğaltılmış ve dizilenmiştir. APOBEC 3G/F hipermutasyonları HIVdb-Stanford algoritması kullanılarak tanımlanmıştır. Çalışma grubunda yer alan hastaların %2.5 (13/515)'inde HIV-1 pol geni dizilerinde APOBEC 3G/F hipermutasyonları saptanmıştır. Hastalarımızda sıklıkla HIV-1 pol geninin RT kangalında ve 3G motifi olarak belirlenen hipermutasyonlar, tedavi-naif hastalarda %2 (8/394), HAART tedavisi altındaki hastalarda ise %4.1 (5/121) oranında tespit edilmiştir. HIV-1 enfeksiyonlarında APOBEC 3G/F hipermutasyonlarının ortaya çıkarılması, HIV-1 enfeksiyonlarının doğasını ve sonuçlarını kavramamıza yardımcı olabilir. Öte yandan APOBEC 3G/F hipermutasyon motif ve sıklığının CD4+ T lenfositlerden elde edilecek HIV-1 proviral DNA örneklerinde araştırılması ve ülkemizde dolaşımda bulunan HIV-1 alttipleri ile ilgisinin ortaya çıkarılması yararlı olabilir.
Anahtar sözcükler: HIV; insan immün yetmezlik virusu; APOBEC3; hipermutasyon; vif; Türkiye.
ABSTRACT
Host genetic factors may play an effective role on the human immunodeficiency virus (HIV) pathogenesis. APOBEC3 (apolipoprotein B mRNA editing enzyme catalytic polypeptide like-3) proteins are cellular antiviral proteins which inhibits HIV replication in the absence of vif (virion infectivity factor). In this study, we aimed to determine the APOBEC 3G/F hypermutations in HIV-1 strains isolated in Turkey. A total of 515 HIV-1 infected patients between June 2009 - February 2012 were included in the study. Three hundred ninety four cases were newly diagnosed antiretroviral-naive patients [349 male, 45 female; medain age (range): 37.1 (2-69) years; median CD4+ T-cell count (range): 340 (1-1660) mm3; median HIV-RNA load (range): 5.76 + E5 (8.7 + E2-9.4 + E6) IU/ml] and 121 were under HAART therapy [99 male, 22 female; median age (range): 40.7 (20-70) years; median CD4+ T-cell count (range): 195 (6-720) mm3; median HIV-RNA load (range): 5.4 + E5 (1.37 + E3-1.07 + E7) IU/ml]. APOBEC 3G/F hypermutations in HIV-1 pol sequences (reverse transcriptase; codons 41-238 and protease; codons 1-99) analysed by nested RT-PCR and direct sequencing techniques. APOBEC 3G/F hypermutations have been determined by using of HIVdb-Stanford algorithm. The prevalence of overall APOBEC 3G/F hypermutations was 2.5% (13/515) in HIV-1 pol gene sequences in study group, and the rates were 2% (8/394) and 4.1% (5/121) in antiretroviral naive and treatment groups, respectively. However, the location and marker hypermutations of determined APOBEC in the HIV-1 pol gene sequences were RT and 3G in the Turkish patients. The hypermutated HIV-1 strains identified in HIV-1 infected patients may facilitate our understanding the nature and the consequences of HIV-1 infections. Moreover, investigations of the motif and frequency of APOBEC 3G/F hypermutations in HIV-1 proviral DNA samples and understanding their relationships with HIV-1 subtypes in Turkish patients would be beneficial.
Key words: HIV; human immunodeficiency virus; APOBEC3; hypermutation; vif; Turkey.
Geliş Tarihi (Received): 15.12.2012 • Kabul Ediliş Tarihi (Accepted): 20.03.2013
GİRİŞ
Bağışıklık sisteminin CD4+ T lenfosit, makrofaj ve dendritik hücreleri gibi temel hücrelerini enfekte eden insan immün yetmezlik virusu (HIV), immün sistemin çökmesiyle sonuçlanan edinilmiş immün yetmezlik sendromuna (AIDS) neden olmaktadır. Zarflı, ikozahedral simetrili, pozitif tek iplikli diploid RNA içeren bir retrovirus olan HIV'ın iki genotipi (HIV-1 ve HIV-2) mevcuttur. Ters transkriptaz (RT) ve integraz enzimleriyle replike olan HIV, enzim ve viral partikülleri sentezleyen gag, pol ve env yapısal genleri, transkripsiyon ve sonrası basamakları düzenleyen ve yayılmayı sağlayan tat ve rev regülatör proteinleri ve vif, vpu, vpr ve nef düzenleyici/aksesuvar proteinleri ile viral patogenezde etkili genetik faktörlere sahiptir1.
Bazı konak genetik faktörleri HIV enfeksiyonu patogenezi üzerinde etkili olmakta ve HIV kliniğinde iyi gidişi olumlu etkileyebilmektedir. Bunlara; APOBEC (apolipoprotein B mRNA editing enzyme catalytic polypeptide-like 3), langerin, tetherin, kemokin reseptör 5 (CCR5) ve HLA B27 gibi doğal bağışıklık sistemi üyeleri örnek olarak verilebilir. Son 10 yılda, HIV-1 enfeksiyonlarında, viral ve konak genetik faktörlerinin karşılıklı etkileşimlerinin yoğun araştırma konusu olduğu gözlenmektedir2. İlk kez 2002 yılında tanımlanan APOBEC3 gen ailesi yedi proteinden (A, B, C, DE, F, G, H) oluşmakta ve virusun replikasyon aşamasında yeni oluşan viral cDNA'yı etkileyerek HIV-1'in enfektivitesini sınırlamaktadır2,3. Buna karşın virus kodladığı vif proteini (virion infectivity factor) ile APOBEC3'ü interfere ederek hücrenin antiviral savunmasından kaçmaktadır. Vif proteini ile etkileşim içinde bulunan APOBEC3 proteinleri vif'in fonksiyonel olmadığı ya da defektif olduğu durumlarda etkin olabilmektedir2,3. HIV'ın genom organizasyonunda vif geni, merkezde ve pol geninin 3' ucuyla üst üste çakışır pozisyondadır.
HIV'ın ters transkripsiyonu sırasında, negatif DNA ipliğindeki CC dinükleotidlerinde, sitozin deaminasyonu sonucunda, pozitif RNA; 5'-GG-3' Æ pozitif DNA; 5'-AG-3' değişimi gerçekleşir. Hipermutasyon olarak tanımlanan bu değişim karakteristik bir APOBEC3 motifidir. Ayrıca G Æ A motifi dışında G Æ non A hipermutasyon motifleri de söz konusudur4. Vif - APOBEC3 etkileşimi açısından bakıldığında APOBEC3G ve APOBEC3F (APOBEC 3G/F) en potent ve antiviral aktiviteleri en iyi bilinen HIV-1 inhibitörleridir3. Bu hücresel defansı HIV, vif aksesuvar proteini sayesinde APOBEC3 protein degradasyonu yoluyla dengelemeye ve nötralize etmeye çalışır. Ancak HIV-1 ile enfekte bireylerde nötralizasyonun etkinliği ve hipermutasyon oluşma sıklığı oldukça değişkendir. APOBEC3 proteinleri ve vif arasındaki denge HIV-1 enfeksiyonunun sonuçları konusunda kritik önemde olabilir5.
G Æ A hipermutasyon motifi ilk kez 1991 yılında, HIV-1'in in vitro çoğaltılması sırasında, env geni dizilerinde tanımlanmıştır6. Hipermutasyonla sonuçlanan nükleotid yer değiştirmelerinin lokal dinükleotid içeriğine bağlı olduğu ve bunun azalan sırayla, GÆA > GÆG > GÆT~ GÆC olarak gerçekleştiği gösterilmiştir6. HIV-1 pol geni dizileri üzerinde yapılan çalışmalara göre yüksek aktif antiretroviral tedavi (HAART) altındaki bireylerin > %9'unda hipermutant proviral DNA'lar saptanmaktadır3,7. Günümüzde HIV-1 dizilerinde APOBEC 3G/F hipermutasyonları biyoinformatik araçlarla rutin olarak taranabilmektedir. HIV-1 dizileri dışında, ters transkriptaz (RT) enzimi ile retrovirus replikasyon özellikleri taşıyan hepatit B virusu (HBV) da hipermutasyonlar yönünden analiz edilebilmektedir8. Kronik HBV taşıyıcısı sekiz hastanın serum örneklerinden elde edilen 92 parsiyel ve tüm HBV genomu dizilerinde iki hastada GÆA hipermutasyon motifi bildirilmiştir9.
Bu çalışmada ülkemizde ilk kez, izole edilen HIV-1 pol geni dizilerinde APOBEC 3G/F hipermutasyonlarının sunulması amaçlanmıştır.
GEREÇ ve YÖNTEM
Çalışma Grubu
Bu araştırma, Kocaeli Üniversitesi Klinik Araştırma Etik Kurulu'nun onayı ile gerçekleştirildi (Protokol: KOU KAEK 2013/45; karar no: 6/6). Çalışmaya, Haziran 2009-Şubat 2012 tarihleri arasında, yeni tanı almış ve primer antiretroviral (ART) ilaç direnci analizi için başvuran 394 hasta [349 erkek, 45 kadın; ortanca yaş (aralık): 37.1 (2-69) yıl; ortanca CD4+ T-hücre sayısı (aralık): 340 (1-1660) mm3; ortanca HIV-RNA yükü (aralık): 5.76 + E5 (8.7 + E2-9.4 + E6) IU/ml] ile HAART tedavisi altında ve HIV-1 RNA yüklerindeki artış sonrasında sekonder ilaç direnci analizi için başvuran 121 hasta [99 erkek, 22 kadın; ortanca yaş (aralık): 40.7 (20-70) yıl; ortanca CD4+ T-hücre sayısı (aralık): 195 (6-720) mm3; ortanca HIV-RNA yükü (aralık): 5.4 + E5 (1.37 + E3-1.07 + E7) IU/ml] olmak üzere toplam 515 HIV-1 pozitif hasta dahil edildi.
Hastaların tanı ve takipleri, HIV-1 ART ilaç direnci analizi talebinde bulunan hastanelerde yapıldı. Hastalarda HIV-1 enfeksiyon tanısı ve HAART tedavisi EACS (European AIDS Clinical Society) ve DHHS (U.S. Department of Health and Human Services; http://aidsinfo.nih.gov/Guidelines) kılavuzlarına göre yapıldı10. HIV-1 enfeksiyonunun klinik evrelemesi, CDC (Center for Diseases Control and Prevention) önerilerine göre CD4+ T lenfosit sayısı esas alınarak yapıldı11. Tedavi öncesi alınan EDTA'lı kan örneklerinin plazmaları ayrıldı ve çalışılıncaya kadar -80°C'de saklandı. Hastalarda tekrarlı olarak pozitif saptanan anti-HIV-1/2 antikor sonuçları Western blot (WB) yöntemi ile (DIA PRO, HIV-1 LIA, Diagnostic Bioprobes Srl, İtalya) doğrulandı. Anti-HIV-1/2 antikor ve doğrulama testleri sırasında hastaların kimliği gizlendi ve örnekler yeniden verilen kodlarla çalışıldı.
HIV-1 RT-PCR ve pol Geni Dizilemesi
HIV-1 pol geninin RT (kodon 41-238) ve proteaz (kodon 1-99) kangalları nested ters transkriptaz polimeraz zincir reaksiyonu (RT-PCR) ile çoğaltıldı ve dizilendi. Bu amaçla The French ANRS (National Agency for AIDS Research) AC11 Direnç Grubu'nun PCR ve dizileme algoritmasından yararlanıldı (www.hivfrenchresistance.org). Kullanılan primerler Tablo I'de gösterildi. RT-PCR koşulları 95°C - 10 dakika, ardından 45 döngü 95°C - 45 saniye, 55°C - 45 saniye ve 72°C - 45 saniye olarak uygulandı. HIV-1 cDNA sentezi M-MuLV ters transkriptaz enziminin kullanıldığı First Strand cDNA Synthesis Kit (Thermo Scientific Inc, Fermentas, Litvanya) ile gerçekleştirildi. PCR ürünleri, DNA Engine peltier termal döngü platformunda (BioRad Laboratories, ABD), 95°C - 10 dakika ön denatürasyon, 45 döngü 95°C - 45 saniye 55°C - 45 saniye ve 72°C - 45 saniye koşullarında elde edildi. Primerler, PCR için 0.2 uM, dizileme için 0.3 uM final konsantrasyonunda kullanıldı. Tüm PCR ürünleri High Pure PCR Product Purification Kit (Roche Diagnostics GmbH, Almanya) ile saflaştırıldı ve ürünler ABI PRISM 310 platformunda DYEnamic ET Terminator Cycle Sequencing Kit (Amersham Pharmacia Biotech Inc., ABD) kullanılarak dizilendi. Direkt dizileme için kullanılan PCR protokolü; 35 döngü 95°C -20 saniye, 50°C - 25 saniye ve finalde 60°C - 2 dakika olarak gerçekleşti. Elektroferogramlar Vector NTI v5.1 (InforMax, Invitrogen, Life Science Software, ABD) programı aracılığıyla elde edildi.
HIV-1 Alttip, Antiretroviral İlaç Direnci ve APOBEC 3G/F Hipermutasyon Analizi
HIV-1 pol geni dizilerinin alttiplendirmesinde ve ART ilaç direnci mutasyonlarının belirlenmesinde HIVdb - Stanford databazı (www.hivdb.stanford.edu, v6.2.0, Mayıs 2012) kullanıldı. Ancak klinik önemi bulunan ART ilaç direnci mutasyonlarının tanımlanmasında Dünya Sağlık Örgütü'nün 2009 "ilaç direnci mutasyon sürveyansı" (SDRM) kriteri esas alındı12. HIVdb - Stanford algoritması aynı zamanda bilinmeyen (fasta formatında) HIV-1 DNA dizilerindeki APOBEC 3G/F hipermutasyonlarını gen bölgesine ve aminoasit pozisyonlarına göre tanımlayabilmektedir. APOBEC 3G/F hipermutasyonları, rutin HIV-1 ilaç direnci analizi için merkezimize başvuran HIV-1 ile enfekte hastalarda, HIVdb - Stanford algoritması ile saptandı.
APOBEC 3G/F hipermutasyon analizi, HIVdb - Stanford databazının kullandığı dizi kalite kontrol mekanizmalarından biridir ve sürekli güncellenmektedir (son güncelleme; Ocak 2011, http://hivdb.stanford.edu/pages/news/index.html). HIVdb - Stanford algoritması, bilinmeyen HIV-1 pol geni dizilerindeki hipermutasyonları, kullandığı referans konsensus HIV-1 alttip B dizileri ile karşılaştırarak tanımlamaktadır. Karşılaştırmada hipermutasyonlar, referans APOBEC belirteçleri esas alınarak (proteaz bölgesinde 11 adet 3G, 4 adet 3F belirteci; RT bölgesinde 19 adet 3G ve 20 adet 3F belirteci bulunur; ayrıca 3G/F bölgelerinde belirteç olan ART direnci mutasyonları da mevcuttur. Bunlar; proteaz bölgesi için; D30N, G48R, G73S ve RT bölgesi için; M41I, D67N, M184I ve G190R mutasyonları olarak tanımlanmıştır) ROC (receiver-operator curve) eğrilerinin kullanılması ile oluşturulmuş bir kontrol dataseti ile değerlendirilmektedir. Bu dataseti, HIV-1 pozitif ve ART naif 6000'den fazla bireyden elde edilmiş proteaz ve RT dizilerinden oluşmaktadır. HIV-1 pol geninde ilaç direnci analizinin hedef bölgeleri olan proteaz (kodon 1-99) ve RT (kodon 1-240) kangalları triptofan (Trp; TGG) kodlayan 9 adet yüksek korunmuş kodon içermektedir. Trp kodonu en azından bir, potansiyel olarak iki G Æ A substitusyon hedef bölgesi içermekte ve proteinin inaktivasyonuna yol açan stop kodon (TAG, TGA ya da TAA) oluşumuna neden olmaktadır. Bu nedenle HIVdb - Stanford databazında HIV-1 proteaz ve RT ilaç direnci hedef bölgesindeki korunmuş Trp bölgeleri, APOBEC 3G/F hipermutasyonları için indikatör bölgeler olarak kullanılmaktadır13.
BULGULAR
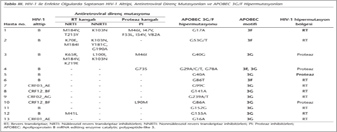
Çalışma grubunda yer alan hastaların %2.5 (13/515)'inde, HIV-1 pol geni dizilerinde APOBEC 3G/F hipermutasyonları saptanmıştır. Klinik ve laboratuvar bulguları Tablo II'de verilen bu hastaların hepsi erkek olup ortanca yaş 41.1 yıl; ortanca CD4+ T-hücre sayısı 209.9 mm3 ve ortanca HIV-RNA yükü 1.08 + E5 IU/ml'dir.
Hipermutasyonlar, ART naif hastalarda %2 (8/394) ve HAART tedavisi altındaki hastalarda %4.1 (5/121) oranında belirlenmiştir. HIV-1 pol geni proteaz kangalında sırasıyla 17., 29., 40., 78. ve 86.; RT kangalında sırasıyla 16., 40., 53., 86., 99., 141., 152., 155. ve 239. nükleotid pozisyonlarında APOBEC 3G/F hipermutasyonları saptanmıştır. HIVdb-Stanford databazının kullandığı APOBEC belirteçlerine göre hipermutasyonların 10'u 3G ve 4'ü 3F motifi olarak tanımlanmıştır. APOBEC 3G/F hipermutasyonları, HIV-1'in pol genindeki lokasyonuna göre 9'u RT; 3'ü ise proteaz kangalında tespit edilmiştir. Ayrıca saptanan hipermutasyonlara 2 adet belirteç ART direnci mutasyonunun; M184I (RT) ve G73S (proteaz) eşlik ettiği belirlenmiştir.
APOBEC 3G/F hipermutasyonları saptananan hastalarda HIV-1 alttip B, CRF01_AE, CRF02_AG, CRF03_AE ve CRF12_BF enfeksiyon etkeni olarak tanımlanmıştır. ART naif iki hastada ve HAART tedavisi altındaki dört hastada çeşitli nükleozid revers transkriptaz inhibitörleri (NRTI), nonnükleozid revers transkriptaz inhibitörleri (NNRTI) ve proteaz inhibitörlerine (PI) sırasıyla primer ve sekonder dirençten sorumlu mutasyonlar belirlenmiştir. Hastalarda saptanan HIV-1 alttip, antiretroviral direnç mutasyonları ve APOBEC 3G/F hipermutasyonları Tablo III'te görülmektedir.
TARTIŞMA
Bu çalışmada sunduğumuz APOBEC 3G/F hipermutasyonları, plazmadan izole edilen HIV-1 dizilerinde tespit edilmiştir. Bu durum, hipermutant HIV-1 kökenlerinin hastalarda bloklanmadan dolaşıma geçmiş olabileceğini gösterebilir. Kieefer ve arkadaşlarının7 HAART tedavisi altındaki dokuz hastada (üç ay boyunca tekrarlanan örneklerinde viral yükün < 50 kopya/ml saptandığı hastalar) hem hücresel (proviral) hem de plazma HIV-1 izolatlarının pol geni dizilerinde APOBEC3 hipermutasyonlarını araştırdığı çalışmasında, RT kangalında %11, proteaz kangalında %13.5 oranında hipermutasyon saptanmış; hastaların proviral HIV-1 DNA'larında hipermutasyonların bulunduğu ancak plazmada, dolaşımda bulunan HIV-1 izolatlarında bulunmadığı bildirilmiştir. Bu sonuçlar, APOBEC 3G/F hipermutasyonlarının etkili HAART tedavilerinde HIV-1 proviral DNA'da arşivlendiğini ve dolaşıma geçmeyebileceklerini gösterebilir7.
Çalışmamıza göre hastalarımızda APOBEC hipermutasyonları, HIVdb-Stanford databazının kullandığı belirteçlere göre HIV-1 pol geninin sıklıkla RT kangalında ve 3G motifi olarak karşımıza çıkmaktadır. APOBEC hipermutasyonlarının lokasyon ve motif dağılımı, HIV-1 sürveyansında önemli olabilir. Ayrıca Tablo III'te görüldüğü gibi mutasyonel belirteçlerden ART direnci ile ilişkili olanların (RT: M184I; proteaz: G73S) yanında başka direnç mutasyonları da bulunmaktadır (RT: M41L, K65R, K70E, L100I, K103N, Y181C, M184V, G190A, T215Y ve K219E; proteaz: M46I, I47V, F53L, I54V, V82A ve L90M). HIV ilaç direncinin bulaştırılabilir olduğu dikkate alındığında bu bulguların HIV-1 sürveyansında ve ayrıca databaz güncellemelerinde yeri olabilir.
APOBEC 3G/F hipermutasyonları saptanan hastaların demografik, klinik ve laboratuvar bulguları ülkemizde daha önce yapılmış çalışmalardaki bulgularla paralellik göstermektedir (Tablo II, III). Ülkemizde HIV-1 pozitif bireylerin belirgin bir oranda (%81) erkek cinsiyetinde olduğu ve heteroseksüel ilişkinin en yaygın bulaş yolu (%64) olduğu anlaşılmaktadır. Filogenetik analizler, alttip B'nin en büyük HIV-1 grubunu oluşturduğunu, ancak belirgin bir şekilde rekombinant suşların da (CRF02_AG, CRF12_BF, CRF03_AB ve CRF01_AE) dolaşımda bulunduğunu göstermektedir. Öte yandan ülkemizde HIV-1 ile ilgili yapılmış az sayıdaki moleküler çalışmada ART direnci mutasyonları sıklıkla ART naif bireylerde analiz edilmiştir14,15,16,17,18. Bu çalışmada HAART tedavisi altındaki HIV-1 ile enfekte hastalarda ART direnci mutasyonlarının saptandığı ve bu mutasyonların ART naif bireylerde saptanmayan paternler içerdiği (sırasıyla NRTI: 65., 70. ve 184. aminoasit pozisyonu; NNRTI: 100., 181. ve 190. aminoasit pozisyonu; PI: 46., 53. ve 90. aminoasit pozisyonu) görülmektedir (Tablo III).
Vif - APOBEC3 etkileşiminin farmakolojik hedef olabileceğine yönelik çalışmalar mevcuttur3. Bu anlamda vif antagonisti yeni sınıf antiviral ilaçlarla HIV-1'in enfektivitesinin azaltılabileceği düşünülmektedir19. Ancak Gandhi ve arkadaşları20, HAART tedavisindeki bireylerden elde edilen 978 bağımsız HIV-1 proviral DNA örneğinde, env geni direkt dizileme analizlerine göre APOBEC3G/F hipermutasyon sıklığını yaklaşık %20 oranında bildirmektedir. Janini ve arkadaşları21, periferik mononükleer kan hücrelerinde pol geni proteaz kangalı dizi analizinde %43 oranında hipermutant HIV-1 kökeni tanımlamışlardır. Bildirilen bu yüksek oranlar, APOBEC3G/F aktivitesinin tek başına viremiyi kontrol edebilecek bir mekanizma olmadığını, muhtemelen sitotoksik T lenfosit yanıtının da içinde bulunduğu diğer viral ve konak faktörlerinin bir kombinasyonu ile HIV-1 baskılanmasının düzenlendiğini gösterebilir7. Öte yandan APOBEC 3G/F hipermutasyonları, HIV-1 ters transkripsiyonunda oluşturduğu defekt ile antiretroviral aktivite göstermektedir. Ancak HIV-1 ile ilgili son zamanlarda ortaya çıkan kanıtlar, hipermutasyonların, ilaç direncine ve immün sistemden kaçışa yol açan genetik değişimlere yol açabileceğini göstermektedir4.
Hepatit B virusunun genom organizasyonunda, pol geni HBsAg'yi kodlayan S geni ile üst üste çakışmaktadır. Bu nedenle HBV oral antiviral ilaç direnci analizlerinde HBsAg "escape" mutasyonların da analiz edilmesi mümkün olabilmektedir22,23,24. HIV'ın genom organizasyonunda da benzer bir özellik bulunmakta ve vif gen bölgesi, pol geninin 3' ucuyla üst üste çakışmaktadır2. Bu genomik özellik vif dizilerinin analizini kolaylaştırabilir. HIV-1 alttiplendirmesi ya da antiretroviral ilaç direncinin rutin olarak analiz edildiği durumlarda elde edilecek olan HIV-1 pol geni dizilerinde, APOBEC 3G/F hipermutasyonları da analiz edilebilir.
HIV-1 dizilerinin moleküler analizleri virusun tanısında, patogenezinde, bulaşında, klinik yönetiminde ve aşı geliştirme çabalarında önem taşımaktadır25. Ülkemizde HIV/AIDS olgusunun bildirimi ilk kez 1985 yılında yapılmasına rağmen26, HIV-1 ile ilgili moleküler temelli çalışmalar az sayıdadır ve bu nedenle HIV-1 alttiplerinin moleküler karakterleri hakkında bilgilerimiz kısıtlıdır. HIV-1 enfeksiyonlarında APOBEC 3G/F hipermutasyonlarının ortaya çıkarılması, HIV-1 enfeksiyonlarının doğasını ve sonuçlarını kavrayışımıza katkı yapabilir. Öte yandan APOBEC 3G/F hipermutasyon motif ve sıklığının CD4+ T lenfositlerden elde edilecek HIV-1 proviral DNA örneklerinde araştırılması ve ülkemizde dolaşımda bulunan HIV-1 alttipleri ile ilgisinin ortaya çıkarılması yararlı olabilir.
KAYNAKLAR
- Turner BG, Summers MF. Structural biology of HIV. J Mol Biol 1999; 285(1): 1-32.
- Albin JS, Harris RS. Interactions of host APOBEC3 restriction factors with HIV-1 in vivo: implications for therapeutics. Expert Rev Mol Med 2010; 12: e4.
- Wissing S, Galloway NL, Greene WC. HIV-1 vif versus the APOBEC3 cytidine deaminases: an intracellular duel between pathogen and host restriction factors. Mol Aspects Med 2010; 31(5): 383-97.
- Wood N, Bhattacharya T, Keele BF, et al. HIV evolution in early infection: selection pressures, patterns of insertion and deletion, and the impact of APOBEC. PLoS Pathog 2009; 5(5): e1000414.
- Armitage AE, Deforche K, Chang C, et al. APOBEC3G - induced hypermutation of human immunodeficiency virus type - 1 is typically a discrete 'all or nothing'' phenomenon. PLoS Genet 2012; 8(3): e1002550.
- Vartanian JP, Meyerhans A, Asjo B, Wain-Hobson S. Selection, recombination, and GÆA hypermutation of human immunodeficiency virus type 1 genomes. J Virol 1991; 65(4): 1779-88.
- Kieffer TL, Kwon P, Nettles RE, et al. G3A hypermutation in protease and reverse transcriptase regions of human immunodeficiency virus type 1 residing in resting CD4+T cells in vivo. J Virol 2005; 79(3): 1975-80.
- Rose PP, Korber BT. Detecting hypermutations in viral sequences with an emphasis on GÆA hypermutation. Bioinformatics 2000; 16(4): 400-1.
- Günther S, Sommer G, Plikat U, et al. Naturally occurring hepatitis B virus genomes bearing the hallmarks of retroviral GÆA hypermutation. Virology 1997; 235(1): 104-8.
- European AIDS Clinical Society (EACS) Guidelines. Version 6, October 2011. www.europeanaidsclinicalsociety.org.
- Centers for Disease Control and Prevention. Guidelines for national human immunodeficiency virus case surveillance, including monitoring for human immunodeficiency virus infection and acquired immunodeficiency syndrome. MMWR Recomm Rep 1999; 48(RR-13): 1-27, 29-31.
- Bennett DE, Camacho RJ, Otelea D, et al. Drug resistance mutations for surveillance of transmitted HIV-1 drug-resistance: 2009 update. PLoS One 2009; 4(3): e4724.
- Gifford RJ, Rhee SY, Eriksson N, et al. Sequence editing by apolipoprotein B RNA-editing catalytic component-B and epidemiological surveillance of transmitted HIV-1 drug resistance. AIDS 2008; 22(6): 717-25.
- Sayan M, Kumbasar Karaosmanoğlu H, Mete B, et al. Molecular epidemiological analysis of HIV-1 pol gene sequences isolated in Istanbul, Turkey. Mikrobiyol Bul 2013; 47(1): 87-97.
- Akhan S, Sayan M, Sargın F. HIV-1 subtypes and antiretroviral resistance mutations in HBV/HCV coinfected HIV-1 positive patients from two center of Marmara region of Turkey. EASL Monothematic Conference: HIV & The Liver. December 7-8, 2012. London, UK.
- Üser ÜY, Sayan M, İnan D. Antalya bölgesinde HIV-1 subtip dağılımı ve antiretroviral ilaç direnci mutasyonları. Klinik HIV/AIDS Sempozyumu 2012, 23-25 Kasım 2012, Kapadokya.
- Sayan M, Willke A, Özguneş N, Sargın S. Türkiye'de HIV-1 ile enfekte antiretroviral naif hastalarda HIV-1 subtip dağılımı ve primer ilaç direnci mutasyonları. Klinik HIV/AIDS Sempozyumu 2012, 23-25 Kasım 2012, Kapadokya.
- Sayan M, Aydın OA, Mete B, Uzun N, Karaosmanoğlu HK, Gündüz A, Tabak F. HIV-1 primary drug resistance mutations in antiretroviral therapy naive patients in Istanbul, Turkey. J Int AIDS Soc 2012; 15(Suppl 4): 18181.
- Xiao Z, Ehrlich E, Luo K, Xiong Y, Yu XF. Zinc chelation inhibits HIV Vif activity and liberates antiviral function of the cytidine deaminase APOBEC3G. FASEB J 2007; 21(1): 217-22.
- Gandhi SK, Siliciano JD, Bailey JR, et al. Role of APOBEC3G/F-mediated hypermutation in the control of human immunodeficiency virus type 1 in elite suppressors. J Virol 2008; 82(2): 3125-30.
- Janini M, Rogers M, Birx DR, McCutchan FE. Human immunodeficiency virus type 1 DNA sequences genetically damaged by hypermutation are often abundant in patient peripheral blood mononuclear cells and may be generated during near-simultaneous infection and activation of CD4(+) T cells. Virology 2001; 75(17): 7973-86.
- Sayan M, Senturk O, Akhan SC, Hulagu S, Cekmen MB. Monitoring of hepatitis B virus surface antigen escape mutations and concomitantly nucleos(t)ide analog resistance mutations in Turkish patients with chronic hepatitis B. Int J Infect Dis 2010; 14(Suppl 3): e136-41.
- Sayan M, Akhan SC. Antiviral drug-associated potential vaccine-escape hepatitis B virus mutants in Turkish patients with chronic hepatitis B. Int J Infect Dis 2011; 15(10): 722-6.
- Sayan M, Cavdar C, Dogan C. Naturally occurring polymerase and surface genes variants of hepatitis B virus in Turkish hemodialysis patients with chronic hepatitis B. Jpn J Infect Dis 2012; 65(6): 495-501.
- Hemelaar J. The origin and diversity of the HIV-1 pandemic. Trends Mol Med 2011; 25(5): 679-89.
- Alp E, Bozkurt I, Doğanay M. Epidemiological and clinical characteristics of HIV/AIDS patients followed-up in Cappadocia region: 18 years experience. Mikrobiyol Bul 2011; 45(1): 125-36.
İletişim (Correspondence):
Doç. Dr. Murat Sayan,
Kocaeli Üniversitesi Tıp Fakültesi Hastanesi,
Merkez Laboratuvarı, PCR Ünitesi,
Eski İstanbul Yolu, 41380, Kocaeli, Türkiye.
Tel (Phone): +90 262 303 8571,
E-posta (E-mail): sayanmurat@hotmail.com