Tedavi Naif Kronik Hepatit B Olgularında Bazal Antiviral Direncin Araştırılması*
Investigation of Baseline Antiviral Resistance in Treatment-Naive Chronic Hepatitis B Cases
Koray ERGÜNAY1, Evrim KAHRAMANOĞLU AKSOY2, Halis ŞİMŞEK2, Alpaslan ALP1, Burçin ŞENER1, Gonca TATAR2,
Dürdal US1, Gülşen HASÇELİK1
1 Hacettepe Üniversitesi Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Ankara.
1 Hacettepe University Faculty of Medicine, Department of Medical Microbiology, Ankara, Turkey.
2 Hacettepe Üniversitesi Tıp Fakültesi, İç Hastalıkları Anabilim Dalı, Gastroenteroloji Ünitesi, Ankara.
2 Hacettepe University Faculty of Medicine, Department of Internal Medicine, Gastroenterology Unit, Ankara, Turkey.
* Bu çalışma, GlaxoSmithKline-Türkiye tarafından desteklenmiş ve 7. Ulusal Moleküler ve Tanısal Mikrobiyoloji Kongresi (5-8 Haziran 2012, Ankara)'nde poster olarak sunulmuştur.
ÖZET
Hepatit B virusu (HBV) ile kronik olarak enfekte ve herhangi bir tedavi almamış (naif) olguların bir kısmında, nükleozid/nükleotid analoğu (NA) bileşiklere karşı antiviral direnç ile ilişkili mutasyonlar saptanabilmektedir. NA ile antiviral tedavi verilmeden önce yapılacak genotipik direnç analizi, optimal tedavi rejiminin seçimi ve erken dönem klinik direncin önlenmesini mümkün kılabilir. Bu çalışmada, kronik hepatit B (KHB) enfeksiyonu tanısı ile takip edilen ve antiviral tedavi almamış olgularda HBV ilaç direnci mutasyonlarının araştırılması amaçlanmıştır. Çalışmaya, Hacettepe Üniversitesi Tıp Fakültesi, İç Hastalıkları Anabilim Dalı Gastroenteroloji Ünitesinde KHB tanısı almış; hepatit C ve D virusu (HCV) ve insan immün yetmezlik virusu (HIV) koenfeksiyonu olmayan, interferon ya da NA ile tedavi başlanmamış 42 olgu (17 kadın, 25 erkek; yaş aralığı: 18-62 yıl), bilgilendirilmiş onam ile dahil edilmiştir. Olgularda HBV viral yükü, serolojik göstergeler ve karaciğer enzimleri, standardize ticari sistemlerle saptanmıştır. HBV ilaç direnci ile ilişkili mutasyonların belirlenmesi için, viral polimeraz geninde sık kullanılan NA ilaçlara karşı direnç mutasyonlarının yerleştiği yaklaşık 250 nükleotidlik kısım "nested" PCR ile çoğaltılarak amplikonlar doğrudan DNA dizi analizi ile incelenmiştir. Elde edilen diziler çeşitli yazılımlar ve web tabanlı bir sanal fenotiplendirme arabirimi kullanılarak bilinen ilaç direnç mutasyonları açısından değerlendirilmiştir. İncelenen 42 örneğin 30 (%71.4)'unda kısmi diziler elde edilmiş ve tüm olgular genotip D olarak saptanmıştır. Olguların 19 (%63.3)'unda çeşitli nükleotid değişiklikleri izlenmiş; bunlar 8 (%26.6) olguda aminoasit değişikliğine neden olmayan sessiz mutasyonlar, 7 (%23.3) olguda ise önemi bilinmeyen mutasyonlar olarak değerlendirilmiştir. Toplam 3 (%10) olguda ise antiviral direnci ile ilişkili mutasyonlar saptanmıştır. Bu mutasyonlar; bir olguda entekavir + lamivudin (S202G, M204V, L180M, T184N), bir olguda muhtemel lamivudin + adefovir (L180P, A181Q), bir olguda ise muhtemel tenofovir direnci (A194V) şeklinde belirlenmiştir. Mutasyon saptanan tüm olgular HBsAg ve HBeAg pozitif, anti-HBe negatif olup HBV viral yükü 3 x 107 IU/ml'nin üstündedir. İki olguda vertikal bulaş söz konusu iken, bir olguda bulaş yolu belirlenememiştir. Olgulardan takip örneği elde edilmesi mümkün olmadığından, izlenen mutasyon profili, serolojik göstergeler ve klinik gidiş ile ilgili değişiklikler izlenememiştir. Sonuç olarak, merkezimizde antiviral tedavisi almamış KHB'li olgularda dirençle ilişkili mutasyonların varlığı gösterilmiştir. Kronik HBV enfeksiyonlarında genotipik direnç testlerinin tedavi öncesi yeri, klinik takibe katkısı ve maliyet etkinliği, prospektif çalışmalarla açıklık kazanacaktır.
Anahtar sözcükler: Hepatit B; hepatit B virusu; kronik; direnç; tedavi; naif; mutasyon.
ABSTRACT
Naturally-occuring mutations associated with resistance to nucleoside/nucleotide analogues (NA) can be detected in a group of treatment-naive individuals chronically infected with hepatitis B virus (HBV). Genotypic resistance testing prior to the initiation of NA therapy may facilitate the selection of optimal drug regime and help to prevent early emergence of clincal resistance. In this study, presence of resistance mutations in treatment-naive individuals with chronic hepatitis B (CHB) was investigated in Hacettepe University Hospital, a referral center in Ankara province, Turkey. A total of 42 patients (17 female, 25 male; age range: 18-62 years) diagnosed as CHB were enrolled in the study with informed consent. All of the patients were negative for hepatitis C and D viruses and human immunodeficiency virus coinfections, and none had a history of interferon or NA treatment. HBV viral load, HBV markers and hepatic enzymes in patients were determined via standardized commercial assays. For the detection of NA resistance mutations, a partial sequence of approximately 250 nucleotides, harboring the frequently-observed sites for NA resistance was amplified via nested PCR and characterized by direct sequencing of the amplicons. The sequences were handled and interpreted for the presence of mutations via various softwares and a web-based virtual phenotyping tool. Well-characterized sequences were obtained in 30 out of 42 samples (71.4%). All circulating HBV strains were observed as genotype D. Nucleotide variations were detected in 19 individuals (63.5%) that comprise silent mutations without amino acid substitution in 8 (26.6%), mutations with undetermined significance in 7 (23.3%) and mutations associated with NA resistance in 3 (10%) patients. Mutations conferring resistance to entecavir + lamivudine (S202G, M204V, L180M, T184N) were identified in one patient whereas L180P, A181Q and A194V substitutions associated with probable lamivudin + adefovir and tenofovir resistance, respectively, were detected in other patients. All patients with resistance mutations were HBsAg and HBeAg positive, anti-HBe negative and had viral loads exceeding 3 x 107 IU/ml. In two patients, the route for HBV transmission was vertical. Since no follow-up samples were available from individuals with resistance mutations, alterations in serological markers, viral load and mutation patterns could not be monitored. In conclusion, the presence of NA resistance mutations were revealed in treatment-naive CHB cases in a referral hospital in Turkey. The impact and cost-effectivity of detecting naturally-occuring resistance mutations for clinical follow-up prior to the antiviral therapy need to be elucidated by prospective studies.
Key words: Hepatitis B; hepatitis B virus; chronic; resistance; treatment; naive; mutation.
Geliş Tarihi (Received): 30.05.2013 • Kabul Ediliş Tarihi (Accepted): 26.08.2013
GİRİŞ
Hepatit B virus (HBV) enfeksiyonları, etkin bir aşının yaygın olarak uygulanmasına rağmen tüm dünyada 2 milyardan fazla kişiyi etkileyen önemli bir halk sağlığı sorunu olmayı sürdürmektedir1. Küresel HBV prevalansı bölgelere göre farklılık göstermektedir. Yaklaşık %4-5 oranında HBsAg (hepatit B yüzey antijeni) seroprevalansına sahip olan Türkiye, Akdeniz çevresi ve Ortadoğu ülkeleri ile birlikte orta endemik bölgeler içerisinde yer almaktadır2,3. Ülkemizde en sık rastlanan HBV genotipi, genotip D olarak bildirilmektedir4.
HBV ile enfekte kişilerde enfeksiyonun kronikleşmesi; kronik hepatit, karaciğer yetmezliği, siroz ve hepatoselüler karsinom gibi ciddi komplikasyonlara zemin hazırlar1. Kronik hepatit B (KHB) tedavisinde günümüzde interferon/pegile interferon ve çeşitli nükleozid/nükleotid analogları (NA) kullanılmaktadır. Lamivudin, adefovir, entekavir, telbivudin ve tenofovir, Avrupa Birliği ve ülkemizde kullanım lisansı bulunan NA'dır5. Ancak NA'nın kullanımı ile virusun eradikasyonu mümkün olmamakta; viral polimeraz geninin çeşitli bölgelerinde ortaya çıkan mutasyonlar antiviral direncine neden olarak ilacın etkinliğini azaltmaktadır6. KHB tanısı ile takip edilen hastalarda, antiviral ilaç direnci ile ilişkili mutasyonların erken dönemde tespiti önemlidir. Bu sayede en uygun tedavi protokolü belirlenerek gereksiz ilaç kullanımının getireceği toksisite ve ekonomik kayıpların önlenmesi; ayrıca ciddi komplikasyonların gelişme insidansının azaltılması mümkündür5,6,7. HBV ile enfekte ve herhangi bir tedavi almamış (naif) olguların bir kısmında da antiviral direnç ile ilişkili mutasyonlar saptanabilmekte, mutant virusları taşıyan kişilerde bazı ilaçların etkinliğinin azaldığı bilinmektedir6. Muhtemel bir antiviral tedavi uygulamasına başlanmadan yapılacak direnç analizi, optimal tedavi rejiminin seçimi açısından yol gösterici olmaktadır5,6,7. Bu çalışmada, Hacettepe Üniversitesi Tıp Fakültesi Hastanesi'nde kronik HBV enfeksiyonu tanısı almış ve antiviral tedavi uygulanmamış olgularda, sık kullanılan antiviral ilaçlara karşı direnç varlığı ve dirençle ilişkili mutasyonların incelenmesi amaçlanmıştır.
GEREÇ ve YÖNTEM
İncelenen Olgular ve HBV Göstergelerinin Saptanması
Hacettepe Üniversitesi Tıp Fakültesi, İç Hastalıkları Anabilim Dalı, Gastroenteroloji Ünitesinde kronik hepatit B tanısı konulan, hepatit C ya da hepatit D koenfeksiyonu olmayan ve antiviral tedavi almamış 42 olgu (17 kadın, 25 erkek; yaş aralığı: 18-62 yıl, yaş ortalaması 32.7 yıl), bilgilendirilmiş onam sonrası çalışmaya dahil edildi. Hastaların rutin takipleri sırasında kan örnekleri alınarak serumları ayrıldı ve çalışılıncaya kadar -80°C'de saklandı. Çalışma protokolü Hacettepe Üniversitesi Etik Kurulu tarafından onaylandı (FON 09/45-188).
Olgularda, örnekleme ile eşzamanlı alanin aminotransferaz (ALT) ve aspartat aminotrasferaz (AST) düzeyleri, HBV serolojik göstergeleri (HBsAg, HBeAg, anti-HBe) ve HBV viral yükü saptandı. Bu amaçla serolojik belirteçlerde kemilüminesans yöntemi (Architect, Abbott, Almanya); ALT-AST düzeylerinde modüler bir otoanalizör (Roche/Hitachi, Japonya) ve HBV-DNA düzeylerinde gerçek zamanlı PCR yöntemi (COBAS AmpliPrep/COBAS TaqMan HBV Test, version 2.0, Roche, ABD) üreticilerin önerileri doğrultusunda uygulandı.
HBV Direnç Mutasyonlarının Saptanması
Hastalara ait serum örneklerinden DNA izolasyonu, ticari bir spin kolon nükleik asit ekstraksiyon kiti olan "High Pure Viral Nucleic Acid Kit" (Roche Diagnostics, Almanya) ile, üreticinin önerileri doğrultusunda yapıldı. Klinikte yaygın olarak kullanılan antivirallere karşı dirençle ilişkili mutasyonların saptanması için HBV polimeraz geninin 470-720. nükleotidleri arasında yer alan yaklaşık 250 baz uzunluğundaki kısmı, iki turlu (nested) PCR yöntemi ile çoğaltıldı8,9. "Nested" PCR ikinci tur ürünleri %2'lik agaroz jel elektroforezine alınarak beklenen büyüklükte amplikonların varlığı doğrulandı. Bant gözlenen amplifikasyon ürünleri, Invisorb Rapid PCR® (Invitek, Almanya) sistemi ile temizlendi. PCR ikinci turda kullanılan sens primer ve "BigDye® Terminator v3.1 Cycle Sequencing Kit" (Applied Biosystems, ABD) kullanılarak dizi analizi reaksiyonu gerçekleştirilen örnekler, ABI PRISM 310 Genetic Analyzer (AppliedBiosystems, ABD) cihazında analiz edildi. Elde edilen diziler Chromas (versiyon 2.13) (Technelysium Pty Ltd, ABD) ve BioEdit (versiyon 7.0.5) (http://www.mbio.ncsu.edu/bioedit/bioedit.html) yazılımları kullanılarak referans diziler ile karşılaştırıldı. Diziler ek olarak internet tabanlı bir sanal fenotiplendirme arabirimi (geno2pheno.HBV, versiyon 2, Genafor, Almanya) kullanılarak değerlendirildi.
BULGULAR
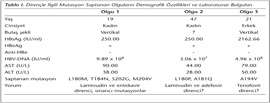
Çalışmamızda incelenen 42 örneğin 30 (%71.4)'unda, PCR ve dizi analizi ile HBV polimeraz genine ait kısmi diziler elde edilmiştir. Tüm suşların HBV genotip D olduğu saptanmıştır. Olguların 19 (19/30, %63.3)'unda incelenen bölgede referans izolatlara göre çeşitli nükleotid değişiklikleri gözlenmiştir. Saptanan değişiklikler; 8 (%26.7) olguda aminoasit değişikliğine neden olmayan sessiz mutasyonlar, 7 (%23.3) olguda ise önemi bilinmeyen mutasyonlar olarak değerlendirilmiştir. Toplam 3 (3/30, %10) olguda antiviral ilaç direnci ile ilişkili mutasyonlar saptanmıştır (Şekil 1). Bunlar; bir olguda entekavir + lamivudin (S202G, M204V, L180M, T184N), bir olguda muhtemel lamivudin + adefovir (L180P, A181Q), bir olguda ise muhtemel tenofovir direnci (A194V) şeklinde izlenmiştir (Tablo I).
TARTIŞMA
HBV ile enfekte kişilerde yapılan çalışmalar, ilaca bağlı bir seçilim baskısı mevcut olmasa da, virusun doğal replikasyonu sırasında dirençle ilişkili mutasyonların ortaya çıkabileceğine işaret etmektedir10,11. Genel olarak, daha önce tedavi almamış kişilerde saptanabilen viral polimeraz bölgesindeki tüm mutasyonların klinik önemi konusunda bilgiler yeterli düzeyde değildir5. Ancak kronik HBV tedavisinde yaygın olarak kullanılan lamivudin ve adefovire karşı tedavi-naif kişilerde izlenen yanıtsızlık ve primer direnç mutasyonları, özellikle bazı NA'nın kullanımı öncesinde genotipik duyarlılık profillerinin ortaya konulmasının klinik yararı olacağını vurgulamaktadır5,10,11,12. Daha önce yaptığımız bir çalışmada, Hacettepe Üniversitesi Hastanesinde, bir yıldan uzun süre NA tedavisi altındaki olgularda direnç mutasyonları araştırılmış ve dizi analizi ile olguların %25.5 (13/51)'inde, lamivudin, entekavir ve adefovir direnci ile ilişkili primer ve onarıcı mutasyonlar saptanmıştır13. Aynı merkezde yapılan bir başka çalışmada ise, HBsAg seroprevalansı ve HBeAg pozitif olguların azalma eğilimi gösterdiği belirtilmiş; HBV ile enfekte kişilerde yaygın bulaş yolunun horizontal geçiş olduğu rapor edilmiştir14. Bu çalışmanın amacı, hiç tedavi almamış kronik HBV olgularında NA direnci ile ilişkili mutasyonların araştırılmasıdır. Çalışmamızda, HBV polimerazını kodlayan ve ilaç direncinden sorumlu birçok mutasyonun ortaya çıktığı bir bölge, yaygın olarak kullanılan bir PCR protokolü ile amplifiye edilmiş ve ürünler dizi analizi yaklaşımı ile karakterize edilmiştir. Dizileri değerlendirilebilen 30 olgunun 3 (%10)'ünde, HBV NA direnci ile ilişkili mutasyonlar tespit edilmiştir (Tablo I).
Ülkemizde değişik hasta gruplarında yapılan incelemelerde, tedavi almamış olgularda değişik sıklıkta ilaç duyarlılığını azaltan çeşitli mutasyonların saptanabildiği ortaya konulmuştur12,15,16,17. Bizim çalışmamızda mutasyon saptanan üç olgunun hepsi HBeAg pozitif ve aktif viral replikasyon izlenen (viral yük > 3 x 107 IU/ml) kişilerdir (Tablo I). Bilindiği gibi, viral replikasyonun yüksek düzeyde devam etmesi, virusun ters transkriptaz enzimi aracılı replikasyonuna bağlı olarak mutant genomların ortaya çıkışını kolaylaştırmaktadır6. Çalışmamızda iki olguda, lamivudin, adefovir ve entekavire azalmış duyarlılık oluşturan iyi tanımlanmış mutasyonlar, bir olguda ise bu mutasyonlara bağlı replikasyon dezavantajını azaltmaya katkıda bulunan onarıcı mutasyon izlenmiştir17,18. Diğer bir olguda ise tenofovir direnci ile ilişkili bir mutasyon gözlenmiştir (Tablo I). Tenofovirin klinik uygulamasında henüz direnç rapor edilmemiş, ancak in vitro koşullarda azalmış duyarlılık oluşturan çeşitli mutasyonlar tanımlanmıştır19,20. Buna karşın ortaya çıkan mutasyonların, virusun replikasyon kapasitesini de önemli düzeyde azalttığı bilinmektedir5,19. Tenofovir tedavisinin, daha önceden lamivudin ya da entekavir uygulanmış ve direnç mutasyonu taşıyan, ayrıca adefovire kısıtlı düzeyde yanıt veren kişilerde de virus replikasyonunu başarılı şekilde baskıladığı rapor edilmektedir21,22,23. Çalışma kapsamında, mutasyon saptanan olgulardan ardışık örneklerin elde edilememiş olması nedeniyle, saptanan nükleotid değişimleri, serolojik belirteçler ve virus replikasyonunun takip sırasında göstereceği muhtemel değişiklikleri inceleme imkanımız olmamıştır.
Çalışmamızda, viral genomda hedeflenen bölgede yer alan nükleotid değişikliklerinin karakterizasyonu için PCR ürünlerinden doğrudan DNA dizi analizi yöntemi uygulanmıştır. Bu yöntem, çoğaltılan bölgedeki tüm mutasyonların ortaya konulması avantajına sahiptir ve nükleotid değişikliklerinin saptanması için altın standart olarak kabul edilmektedir. Bununla birlikte viral genomda bulunabilecek tüm mutasyonların doğrudan dizi analizi yöntemi ile saptanabilmesi için, mutant genomların tüm viral popülasyonun en az %20'sini oluşturması gerekmektedir. Alternatif olarak, PCR amplikonlarının klonlanarak yüksek sayıda klonun dizi analizi yaklaşımı uygulanabilir22,23. Günümüzde gittikçe yaygınlık kazanan yeni kuşak dizileme sistemleri de, örnekte bulunan genomların tamamının analizine imkan sağlamakta, böylece enfekte kişilerdeki tüm viral genom havuzu incelenebilmektedir. Ancak bu yaklaşımların, rutin uygulamalar dışında, cihaz ve deneyimli personel gereksinimi nedeniyle tüm merkezlerde gerçekleştirilebilmesi mümkün değildir. Buna rağmen, HBV ve HCV enfeksiyonlarında viral türümsülerin (quasispecies) incelenmesi, patogenez ve klinik gidiş için birçok yeni bilgi sağlamaktadır12.
Sonuç olarak merkezimizde. önceden ilaç tedavisi almamış kronik hepatit B olgularında, birçok NA'ya karşı dirençle ilişkili mutasyonların varlığı gösterilmiştir. Bu durumun hasta takibine etkileri ve tedavi öncesi direnç analizinin optimal şekilde uygulanması ile ilgili hususlar, prospektif çalışmalar sonucunda netlik kazanacaktır.
KAYNAKLAR
- Tran TT, Martin P. Hepatitis B: epidemiology and natural history. Clin Liver Dis 2004; 8(2): 255-66.
- Andre F. Hepatitis B epidemiology in Asia, the Middle East and Africa. Vaccine 2000; 18(S1): 20-2.
- Emekdas G, Cavuslu S, Oncul O, Cigdem A, Armagan A. Trends in hepatitis B and hepatitis C virus among blood donors over 16 years in Turkey. Eur J Epidemiol 2006; 21(4): 299-305.
- Bozdayi AM, Aslan N, Bozdayi G, et al. Molecular epidemiology of hepatitis B, C and D viruses in Turkish patients. Arch Virol 2004; 149(11): 2115-29.
- European Association for the Study of Liver. EASL clinical practice guidelines: management of chronic hepatitis B virus infection. J Hepatol 2012; 57(1): 167-85.
- Shaw T, Bartholomeusz A, Locarnini S. HBV drug resistance: mechanisms, detection and interpretation. J Hepatol 2006; 44(3): 593-606.
- Yeon JE. Technique for the early detection of drug-resistant HBV-DNA during antiviral therapy. Intervirology 2008; 51(S1): 7-10.
- Bozdayi AM, Uzunalimoğlu O, Türkyilmaz AR, et al. YSDD: a novel mutation in HBV DNA polymerase confers clinical resistance to lamivudine. J Viral Hepat 2003; 10(4): 256-65.
- Ogata N, Fujii K, Takigawa S, Nomoto M, Ichida T, Asakura H. Novel patterns of amino acid mutations in the hepatitis B virus polymerase in association with resistance to lamivudine therapy in japanese patients with chronic hepatitis B. J Med Virol 1999; 59(3): 270-6.
- Fung SK, Chae HB, Fontana RJ, et al. Virologic response and resistance to adefovir in patients with chronic hepatitis B. J Hepatol 2006; 44(2): 283-90.
- Margeridon-Thermet S, Shulman NS, Ahmed A, et al. Ultra-deep pyrosequencing of hepatitis B virus quasispecies from nucleoside and nucleotide reverse-transcriptase inhibitor (NRTI)-treated patients and NRTI-naive patients. J Infect Dis 2009; 199(9): 1275-85.
- Yildiz O, Aygen B, Demirtürk N, et al. Lamivudine resistance mutations in patients infected with hepatitis B virus genotype D. World J Gastroenterol 2011; 17(45): 4987-92.
- Aydogan S, Ergünay K, Balaban Y, et al. Detection of resistance mutations in chronic hepatitis B patients receiving antiviral therapy for over one year. Mikrobiyol Bul 2013; 47(3): 472-81.
- Ergunay K, Balaban Y, Cosgun E, et al. Epidemiologic trends in HBV infections at a reference centre in Turkey: an 11-year retrospective analysis. Ann Hepatol 2012; 11(5): 672-8.
- Akarsu M, Sengonul A, Tankurt E, et al. YMDD motif variants in inactive hepatitis B carriers detected by Inno-Lipa HBV DR assay. J Gastroenterol Hepatol 2006; 21(12): 1783-8.
- Tunçbilek S, Köse S, Elaldi A, Akman S. Lamivudine re-sistance in untreated chronic hepatitis B patients in Turkey. Turk J Gastroenterol 2008; 19(2): 99-103.
- Sayan M, Akhan SC, Meric M. Naturally occurring amino-acid substitutions to nucleos(t)ide analogues in treatment naive Turkish patients with chronic hepatitis B. J Viral Hepat 2010; 17(1): 23-7.
- Lok AS, Zoulim F, Locarnini S, et al. Antiviral drug-resistant HBV: standardization of nomenclature and assays and recommendations for management. Hepatology 2007; 46(1): 254-65.
- Qin B, Budeus B, Cao L, et al. The aminoacid substitutions rtP177G and rtF249A in the reverse transcriptase domain of hepatitis B virus polymerase reduce the susceptibility to tenofovir. Antiviral Res 2013; 97(2): 93-100.
- Liu Y, Wang CM, Cheng J, et al. Hepatitis B virus in tenofovir-naive Chinese patients with chronic hepatitis B contains no mutation of rtA194T conferring a reduced tenofovir susceptibility. Chin Med J (Engl) 2009; 122(13): 1585-6.
- Baran B, Soyer OM, Ormeci AC, et al. Efficacy of tenofovir in patients with lamivudine failure is not different from that in nucleoside/nucleotide analogue-naive patients with chronic hepatitis B. Antimicrob Agents Chemother 2013; 57(4): 1790-6.
- Karatayli E, Idilman R, Karatayli SC, et al. Clonal analysis of the quasispecies of antiviral-resistant HBV genomes in patients with entecavir resistance during rescue treatment and successful treatment of entecavir resistance with tenofovir. Antivir Ther 2013; 18(1): 77-85.
- Lavocat F, Deny P, Pichoud C, et al. Similar evolution of hepatitis B virus quasispecies in patients with incomplete adefovir response receiving tenofovir/emtricitabine combination or tenofovir monotherapy. J Hepatol 2013; 59(4): 684-95.
İletişim (Correspondence):
Doç. Dr. Koray Ergünay,
Hacettepe Üniversitesi Tıp Fakültesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
Sıhhiye, 06100 Ankara, Türkiye.
Tel (Phone): +90 312 305 1562/131,
E-posta (E-mail): ekoray@hacettepe.edu.tr