N�kleik
Asit Ekstraksiyonunda Kullanılmak �zere, Gram-Pozitif Bakteriler ve
Mikobakterilerin H�cre Duvarlarının Ortadan
Kaldırılmasında Yeni Bir Y�ntem: Kum Y�ntemi
A
New Method for The Disruption of Cell Walls of Gram-Positive Bacteria and
Mycobacteria On The Point of Nucleic Acid Extraction: Sand Method
Fikret ŞAHİN1, Mehmet KIYAN1, Djursun KARASARTOVA2, M. Kerem �ALGIN3, Shameem AKHTER1,
Buse T�REG�N ATASOY1
1 Ankara �niversitesi Tıp Fak�ltesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Ankara.
1 Ankara University Faculty of Medicine, Department of Medical Microbiology, Ankara, Turkey.
2 Hitit �niversitesi Tıp Fak�ltesi, Tıbbi Mikrobiyoloji Anabilim Dalı, �orum.
2 Hitit University Faculty of Medicine, Department of Medical Microbiology, �orum, Turkey.
3 Ordu �niversitesi Tıp Fak�ltesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Ordu.
3 Ordu University Faculty of Medicine, Department of Medical Microbiology, Ordu, Turkey.
�Z
G�n�m�zde molek�ler y�ntemler, enfeksiyon etkenlerinin hızlı tanısında yaygın olarak kullanılmaktadır. Bu ama�la en sık olarak polimeraz zincir reaksiyonu (PCR) y�ntemi tercih edilmektedir. PCR kullanımında yeterli ve saf DNA veya RNA elde edilmesi �nemlidir. Gram-negatif bakterilerde fenol-kloroform, DNAzol, proteinaz K, cam boncuk, kaynatma gibi farklı DNA ekstraksiyon y�ntemleri başarıyla kullanılmaktadır. H�cre duvarında daha kalın peptidoglikan tabakası olan gram-pozitif bakteriler ile kompleks glikolipid bulunan mikobakteri cinsi bakterilerde ise, DNA ve RNA izolasyonu i�in bu kompleks yapıların ortadan kaldırılması gerekmektedir. Bu ama�la, stafilokok cinsi gram-pozitif bakterilerde lizostafin kullanılarak sferoplast oluşturulması, mikobakterilerde setil-trimetil amonyum brom�r gibi kimyasallar kullanılarak bakteri duvarının tamamen veya kısmen uzaklaştırılması gerekmektedir. Herhangi bir kimyasal ajana gerek duyulmaksızın, bakteri h�cre duvarının mekanik olarak ortadan kaldırılmasının ama�landığı bu �alışmada, ince elenmiş kum partik�lleri kullanılmış ve y�ntem "kum y�ntemi" olarak adlandırılmıştır. DNA ekstraksiyonu amacıyla ince elenmiş kum, k���k partik�llerini kaybetmeyecek şekilde ddH2O ile yıkanmış ve otoklavda sterilize edilmiştir. RNA ekstraksiyonu i�in ddH2O ile yıkanan kum, daha sonra %10 HCl'de (30 dakika) ink�be edilmiş ve otoklavda� sterilize edilmiştir. �alışmada, daha �nce klinik bir �rnekten izole edilen ve tanımlanan metisiline diren�li Staphylococcus aureus (MRSA) suşu, 100 mg hazırlanan kum ve 100 �l Tris-EDTA ��zeltisi i�erisinde, 5 dakika maksimum hızda vortekslenmiş, DNA eldesi i�in proteinaz K ile muamele edilmiş ve sonrasında fenol kloroform-etanol presipitasyon protokol� takip edilmiştir. Kum y�nteminin diğer y�ntemler ile kıyaslanması amacıyla, kum y�nteminde kullanılan miktardaki bakteri lizostafin ile bir saat ink�be edildi ve kum y�nteminde kullanılan miktarda proteinaz K eklenerek DNA ekstraksiyon işlemi tamamlanmıştır. Boncuk y�nteminde steril cam boncuklar kum y�nteminde olduğu şekilde kullanılmıştır. RNA eldesi i�in Mycobacterium tuberculosis H37Rv ATCC 25618, M.tuberculosis H37Ra ATCC 25177 ve M.tuberculosis H37Rv Pasteur Enstit�s� RSKK 598 standart suşları, 100 mg hazırlanan kum ve 20 �l Tris-EDTA ��zeltisi i�erisinde 3-5 dakika maksimum hızda vortekslenmiş ve guanidinyum tiyosiyanat-fenol-kloroform (GTFK) ile klasik RNA ekstraksiyon protokol� tamamlanmıştır. Elde edilen DNA'nın kullanılabilirliği, �alışmada kullanılan MRSA suşundaki stafilokinaz ve enterotoksin genlerine �zg�l primerler kullanılarak PCR ile araştırılmıştır. Kum y�ntemi ile elde edilen RNA'nın kullanılabilirliğini saptamak i�in, �nce cDNA sentezi yapılmış; daha sonra M.tuberculosis atım pompa genlerinden Rv1410c, Rv2333c ve DrrA'ya �zg�l primerler kullanılarak PCR uygulanmıştır. Karşılaştırma amacıyla, kum y�nteminde kullanılan miktardaki mikobakteriye doğrudan GTFK protokol� uygulanmıştır. MRSA suşlarından lizostafin uygulanarak elde edilen DNA ile kum y�ntemi uygulanarak elde edilen DNA'lar agaroz jelde y�r�t�lm�ş, spektrofotometrede miktar ve saflıkları kıyaslanmıştır. Sonu� olarak, yaklaşık aynı miktar ve saflıkta DNA'ların elde edildiği g�zlenmiştir. Kum y�ntemiyle elde edilen DNA'ların herhangi bir inhibit�r ajan i�ermediği, PCR'nin etkin olarak �alışmasıyla g�sterilmiştir. Mikobakteri suşlarından kum y�ntemiyle RNA elde edilebildiği halde, diğer y�ntemlerle RNA elde edilememiştir. Kum y�ntemi kullanılarak elde edilen RNA'ların gerek cDNA sentezinde gerekse bu cDNA'ların kullanıldığı PCR y�nteminde etkin olarak �alıştığı g�sterilmiştir. �alışmamızda tanımlanan kum y�ntemi ile n�kleotid eldesi zor olan sert ve kompleks h�cre duvarına sahip bakterilerden, saf ve yeterli miktarda DNA ve RNA elde edildiği belirlenmiştir. Sonu� olarak bu y�ntemin, pahalı sayılabilen lizostafin veya farklı kimyasalların yerine herhangi bir masrafı olmayan kumun kullanılması sayesinde ekstraksiyon maliyetini d�ş�receği, ayrıca DNA ekstraksiyon s�resini kısaltması a�ısından avantajlı olacağı d�ş�n�lm�şt�r.
Anahtar s�zc�kler: DNA; RNA; ekstraksiyon; mikobakteri; stafilokok; kum y�ntemi.
ABSTRACT
Nowadays molecular methods are widely used in the rapid diagnosis of infectious agents. Polymerase chain reaction (PCR) is the most preferred method for this purpose. Obtaining sufficient and pure DNA or RNA is important for the PCR. Different DNA extraction protocols such as phenol-chloroform, proteinase K, glass beads and boiling have been used successfully for DNA isolation from gram-negative bacteria. However since gram-positive bacteria have a thicker layer of peptidoglycan and mycobacteria have complex glycolipids in their cell walls, for the isolation of DNA or RNA from these microorganisms, the complex cell wall structure must be eliminated. For this purpose, the bacterial cell wall must be completely or partially removed forming sferoblast using lysostaphin in the Staphylococcus genus as gram-positive bacteria and using a chemical like cetyltrimethyl ammonium bromide for the Mycobacterium genus. In this study, we planned to use sand particles for the mechanical elimination of the cell wall without any need for chemicals and we called this procedure as "sand method". For the purpose of DNA extraction, the fine-grained sand was washed with ddH2O without losing small particles and then sterilized by autoclaving. For the purpose of RNA extraction; the sand was washed with ddH2O, incubated for 30 minutes with 10% HCl, and then autoclaved. A methicillin-resistant Staphylococcus aureus (MRSA) strain previously isolated and identified from a clinical specimen was mixed in 100 �l Tris-EDTA buffer with 100 mg sand. The mixture of bacteria and sand was vortexed at the maximum speed for 5 minutes. The MRSA-sand mix was treated with proteinase K and phenol-chloroform, and ethanol precipitation protocol was then followed for obtaining DNA. For comparison of the sand method with the other methods, the same amount of bacteria used in the sand method was incubated for one hour with lysostaphin, and then the proteinase K DNA extraction method were completed in the same way used in the sand method. For obtaining RNA from M.tuberculosis H37Rv ATCC 25618, M.tuberculosis H37Ra ATCC 25177 and M.tuberculosis H37Rv Pasteur Institute RSKK 598 standard strains, bacteria were dissolved in 20 �l Tris-EDTA buffer with 100 mg sand. The mixture of bacteria and sand was vortexed at the maximum speed for 5 minutes. After that, the classic RNA extraction protocol using guanidinium thiocyanate-phenol-chloroform (GTPC) was completed. To investigate the usefulness of the obtained DNA, a PCR was performed with specific primers for staphylokinase and enterotoxin genes that were shown in the genome of the chosen MRSA strains from our previous studies. To investigate the usefulness of the obtained RNA from the sand method; first cDNA synthesis is completed. The PCR efficiency was then tested using primers specific to the efflux pump genes of M.tuberculosis including Rv1410c, Rv2333c, and DrrA genes. To compare the effect of the sand method, GTPC protocol was applied in the same amount of mycobacteria without the sand treatment. The DNA obtained from MRSA with the application of lysostaphin and the DNA obtained from MRSA by the sand method were run in agarose gel electrophoresis. The amount and purity of DNAs were measured with a spectrophotometer. The same amount and purity of the DNAs were approximately the same in both of the extraction methods. The existence of non-inhibitors of DNA in the sand method was shown with the PCR, which have worked efficiently with the DNAs obtained from the sand method. RNA was obtained efficiently from the Mycobacterium strains by the sand method, but no RNA could be obtained from the mycobacteria with the other methods. It was shown that the RNA obtained using the sand method worked effectively in both cDNA synthesis and PCR in which synthesized cDNA was used. The sand method described in the study worked effectively to obtain sufficient amount of pure DNA and RNA from the bacteria containing rigid cell walls that are difficult to obtain the nucleotide. It was concluded that, using the sand method instead of relatively expensive lysostaphin or other chemicals, has important advantages such as decreasing the cost and the shortening of the DNA extraction period.
Keywords: DNA; RNA; extraction; mycobacteria; staphylococci; sand method.
Geliş Tarihi (Received): 13.10.2015 • Kabul Ediliş Tarihi (Accepted): 10.12.2015
GİRİŞ
Enfeksiyon hastalıklarının tanısında mikroorganizmanın izolasyon ve tanımlanması altın standart olmasına rağmen, etkenin her zaman �retilememesi ve bakteriyemiler gibi acil tedavi gerektiren durumlar nedeniyle, etken DNA'sının araştırıldığı hızlı tanı y�ntemleri �n plana �ıkmıştır1,2,3. Polimeraz zincir reaksiyonu (PCR) ve ger�ek zamanlı (real-time) PCR gibi, enfeksiyon etkeninin �zg�l DNA veya RNA'sının �oğaltılması ve g�r�nt�lenmesine dayalı y�ntemler, hızlı tanı i�in en etkili y�ntemler olarak bilinmektedir3,4. Bu y�ntemlerin uygulanabilmesi i�in, etken mikroorganizmaya ait DNA'nın saf ve yeterli miktarda elde edilmesi gerekmektedir. Bu nedenle, fenol-kloroform, DNAzol, proteinaz K, termal şok ve kaynatma gibi farklı DNA ekstraksiyon y�ntemleri kullanılmaktadır2,5,6,7,8. Bu y�ntemler, genel olarak gram-negatif bakterilerde başka bir işleme gerek kalmadan uygulanabilirken, kalın peptidoglikan tabaka bulunduran gram-pozitif bakteriler ile h�cre duvarlarında peptidoglikan ve kompleks glikolipid bulunan mikobakterilerde DNA ve RNA izolasyonu i�in bu yapıların ortadan kaldırılması gerekmektedir9. N�kleotid eldesinin zor olduğu enfeksiyon etkenlerinin �nemli kısmını gram-pozitif bakteriler oluşturmaktadır; zira� kalın peptidoglikan tabaka, DNA ekstraksiyonu i�in gerekli h�cre lizisine karşı diren�lidir5,9. Bunun i�in h�cre duvarının ortadan kaldırılarak sferoplast formuna d�n�şmesinin sağlanması gerekmektedir10. Bu işlem yapılmadan standart ekstraksiyon y�ntemlerinin etkili olmadığı bilinmekte ve bu nedenle bakterinin sferoblast formuna d�n�şmesi amacıyla lizostafin enzimi kullanılmaktadır.
Gram-pozitif bakteri h�cre duvar yapısına sahip ve h�cre duvarında bol miktarda kompleks lipidler i�eren mikobakterilerden de DNA ve RNA izolasyonu olduk�a zordur7,11,12. Bu durumlarda setil-trimetil amonyum brom�r (CTAB) gibi h�cre duvarını ortadan kaldıran kimyasallar kullanılmaktadır11. Gerek gram-pozitif bakteriler gerekse mikobakterilerde adı ge�en kimyasal kullanımlarına ek olarak, bakteri duvarının ortadan kaldırılması i�in Fransız basın� (French press), cam boncuk gibi mekanik cihaz ve y�ntemler kullanılmakta; bu y�ntemler de farklı ekipmanlar gerektirmektedir5,13. Bu �alışmada, stafilokok ve mikobakteri cinsi bakterilerden DNA ve RNA ekstraksiyonunda, s�z konusu enzim veya kimyasalları kullanmadan h�cre duvarının mekanik olarak par�alanmasına dayanan "Kum y�ntemi" adını verdiğimiz yeni bir y�ntem geliştirilmiştir.
GERE� ve Y�NTEM
Kum
Doğal olarak kaya ve minerallerden oluşmuş gran�ler yapıda olan kum, akarsu ve deniz sahillerinde daha saf olarak bulunmaktadır. Akarsu kenarında bulunan kum partik�llerinin kenar yapılarının, deniz kumuna g�re daha keskin olması beklenen bir durumdur. Bu nedenle �alışmada, DNA ekstraksiyonu amacıyla akarsu kenarından elde edilen ve b�y�kl�ğ� 0.5-3 mm olacak şekilde elenen ince kum kullanıldı. Kum, i�erisinde kir kalmayıncaya kadar ddH2O ile yıkandı ve otoklavda (121�C'de 20 dakika) sterilize edildi (Resim 1). Elde edilen kum, su i�erisinde 24 saat ink�be edildi ve sonra ddH2O'nun pH değerinde değişiklik olmadığı tespit edildi.
RNA ekstraksiyonu i�in elenmiş kum, aynı şekilde ddH2O ile yıkandıktan sonra %10 HCl ile 37�C'de 30 dakika ink�be edildi, daha sonra tekrar ddH2O ile yıkanarak HCl artığı kalmaması sağlandı ve otoklavda (121�C'de 20 dakika) sterilize edildi.
�alışmada Kullanılan Bakteriler
Bu �alışma, farklı hastalardan izole edilen ve daha �nceki �alışmalarımızda da kullanılan metisiline diren�li Staphylococcus aureus (MRSA) suşları �zerinde y�r�t�ld�14. Gram boyama, katalaz, plazma koag�laz ve DNaz testleri ile tanımlanan S.aureus suşlarının metisilin direnci, oksasilin disk dif�zyon testi ve PCR ile mecA geninin saptanması sonucu belirlendi. Mikrobank saklama t�plerinde -20�C'de saklanan her bir suş i�in birer boncuk alındı ve i�inde 3 ml beyin-kalp inf�zyon buyyonu (Brain Heart Infusion Broth [BHIB]; Difco Laboratories, ABD) bulunan t�plerin i�ine atıldı. T�pler 37�C'de 24 saat ink�be edildikten sonra, %5 koyun kanlı agar besiyerlerine tek koloni d�şecek şekilde ekimler yapıldı. Besiyerleri 37�C'de 24 saat ink�be edildi. Kum y�ntemi ile elde edilen DNA'ların PCR'da �alışabilirliğini belirlemek amacıyla, �nceki �alışmalarımızda bakteriyofajlarla taşındığını tespit ettiğimiz stafilokinaz (SAK) ve enterotoksin (SEK) genlerini bulunduran MRSA suşları kullanıldı.
�alışmada mikobakteri suşları olarak; T�rkiye Halk Sağlığı Kurumu'ndan elde edilen M.tuberculosis H37Rv ATCC 25618, M.tuberculosis H37Ra ATCC 25177 ve M.tuberculosis H37Rv Pasteur Enstit�s� RSKK 598 standart suşları kullanıldı. Mikobakteriler L�wenstein-Jensen (LJ) (Difco, ABD) besiyerinde �retildi.
Kum ile MRSA H�cre Duvarının Par�alanması, DNA Ekstraksiyonu ve DNA Saflığının Belirlenmesi
Her bir suşun kanlı agar besiyerinde 37�C'de bir gecelik k�lt�r�n� takiben alınan bir �ze dolusu bakteri, i�inde 100 mg kum ve 100 �l Tris-EDTA ��zeltisi (20 mM Tris HCl, 140 mM NaCl, 5 mM EDTA [pH 8.0]) (Sigma, Almanya) bulunan ependorf t�p�ne aktarılıp karıştırıldıktan sonra 3200 rpm'de 3-5 dakika şiddetli olarak vortekslendi. Daha sonra 10.000 rpm'de 2 dakika santrif�j edildi ve s�pernatan kısmı atıldıktan sonra �zerine 20 �l proteinaz K (20 mg/ml) (Sigma Almanya) eklendi. 37�C'de 2 saat ink�be edildikten sonra 300 μl fenol (Sigma, Almanya): kloroform (Sigma, Almanya): izoamil alkol (Sigma, Almanya): (25:24:1) eklenerek DNA ekstrakte edildi. Kloroform ile yıkamayı takiben saf etanolde DNA ��kt�r�ld�, %70 ile alkolle yıkanıp ddH2O i�inde ��z�nd�r�ld�. Elde edilen DNA'nın miktarı ve saflığı (A260/A280) Biotek mikropleyt okuyucuda (Synergy HTX Multi-Mode Reader, Biotek, ABD) belirlendi.
Lizostafin İle H�cre Duvarının Par�alanması, DNA Ekstraksiyonu ve DNA Saflığının Belirlenmesi
Kum y�nteminde olduğu gibi, her bir suşun kanlı agar besiyerinde 37�C'de 18 saatlik k�lt�r�n� takiben, bir �ze dolusu koloni, i�inde 100 μl Tris-EDTA ��zeltisi bulunan ependorf t�p�ne aktarıldı, �zerine 50 �l lizostafin (1 mg/ml) (Sigma, Almanya) eklenip 37�C'de 3 saat bekletildikten sonra, �zerine 20 �l proteinaz K (20 mg/ml) konup karıştırıldı. 37�C'de 2 saat ink�be edildikten sonra 300 μl fenol: kloroform: izoamil alkol (25:24:1) eklenerek DNA ekstraksiyon işlemi tamamlandı. Kloroform ile yıkamayı takiben saf etanolde DNA ��kt�r�ld�, %70'lik alkolle yıkanıp ddH2O i�inde ��z�nd�r�ld�. Elde edilen DNA miktarı ve saflığı (A260/A280) Biotek mikropleyt okuyucu ile belirlendi.
Cam Boncuk ile H�cre Duvarının Par�alanması ve DNA Ekstraksiyonu
100 �g 450-600 mikron steril cam boncuk (Sigma, G1277) kum y�nteminde a�ıklandığı şekilde kullanıldı.
M.tuberculosis Suşlarından Kum Y�ntemi ile RNA Ekstraksiyonu ve cDNA Sentezi
LJ besiyerinde �retilen standart mikobakteri suşlarına ait koloniler, besiyeri y�zeyinden bir �ze dolusu olacak şekilde kazınarak toplandı ve 20 �l Tris-EDTA ��zeltisi ve 100 mg kum bulunduran ependorf t�p� i�inde karıştırıldıktan sonra 5 dakika şiddetli olarak (3200 rpm) vortekslendi. �zerine temel olarak, asit guanidinyum tiyosiyanat-fenol-kloroform kullanımına g�re hazırlanmış olan kitin (Invitrogen, ABD) birinci ��zeltisi (guanidinium thiocyanate) eklenip 15 saniye elde şiddetli olarak karıştırıldı ve daha sonra kit protokol�ne g�re RNA elde edildi. Kontrol olarak LJ besiyerinde �remiş koloni direkt olarak RNA ekstraksiyon kitinin birinci ��zeltisi i�erisine konduktan sonra elde şiddetli şekilde karıştırıldı ve kit protokol� takip edildi. Elde edilen RNA'lar -80�C'de saklandı.
RevertAid RT Reverse Transcription Kit (Life Technologies, ABD) protokol�ne g�re her bir suştan elde edilen RNA'lara cDNA sentez işlemi uygulandı. Kısaca; her suş i�in hesaplanan RNA miktarı ve hacmini tamamlamak amacıyla i�inde RNaz bulundurmayan steril distile su toplam hacim 9 �l olacak şekilde karıştırıldı. �zerine 1 �l oligo dNTP (dNTP mix, Thermoscientific, ABD) eklendi, vorteksleme ve santrif�jleme işlemlerinden sonra ısıtma bloğunda 70�C'de 10 dakika bekletildi. 8 �l RT Reaksiyon Mix (Life Technologies, ABD) eklendi, vorteksleme ve santrif�jleme işlemlerinden sonra ısıtma bloğunda 42�C'de bir saat bekletildi. Elde edilen cDNA'lar -20�C'de saklandı.
Primerler ve PCR Y�ntemi
Bu �alışmada kullanılan primerler http://www.ebi.ac.uk/clustalw dizi programı kullanılarak tarafımızdan tasarlandı ve IDT (Integrated DNA Technologies, ABD) firmasında sentez ettirildi. �alışmada kullanılan primerler Tablo I'de g�sterildi. Bu �alışmada, mikobakterilerin atım pompasıyla ilişkili genlerinden olan ve daha �nceki �alışmamızda kullandığımız Rv1410c, Rv2333c ve DrrA genlerine �zg�l primerler kullanıldı15.
Her bir suştan elde edilen DNA veya cDNA'lar kalıp DNA olarak kullanıldı. PCR karışımı; 10x PCR ��zeltisi, 2.5 U Taq DNA polimeraz, 1.5 mM MgCl2, 200 �M dNTP ve 2 �l DNA veya cDNA olacak şekilde hazırlandı. Eppendorf Mastercycler Personal Thermocycler (T10 Thermal Cycler, BioRad, ABD) kullanılarak, 34 d�ng�n�n her biri 94�C'de 30 sn; 60�C'de 45 sn ve 72�C'de 45 sn olacak şekilde uygulandı. �oğaltılmış PCR �r�nleri %1'lik agaroz jelde y�r�t�lerek g�r�nt�lendi.
RNA eldesinin etkinliğinin araştırılması amacıyla, elde edilen cDNA'lara PCR uygulandı. Bu işlemden �nce RNA ekstraksiyonu esnasında olması muhtemel DNA kontaminasyonunu �nlemek i�in, elde edilen RNA ��zeltisi (20-100 ng/�l) i�ersine 1 �l DNaz (2000 u/m M0303-L, New England Biolabs, İngiltere) eklendi; 37�C'de 30 dk ink�be edildikten sonra 75�C'de 10 dk bekletilerek DNaz inaktive edildi. Bu ��zelti kullanılarak yapılan PCR ile DNA kontaminasyonu araştırıldı.
BULGULAR
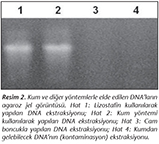
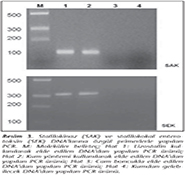
Kum y�nteminin etkinliğinin araştırılması i�in, lizostafin enzimiyle cam boncuk ve kum y�ntemi kullanılarak bakteri h�cre duvarı par�alama işlemi yapıldıktan sonra aynı proteinaz K işlemine tabii tutulmuştur. MRSA suşuna ait DNA ekstraksiyonu sonu�ları karşılaştırıldığında, lizostafin ve kum y�nteminden elde edilen DNA miktarları birbirlerine yakın bulunmuştur. Cam boncuk kullanılan bakteri duvarı par�alama işleminde ise bakteri DNA'sı elde edilememiştir (Resim 2). Bu arada kum y�nteminde kullanılan kumda DNA kontaminasyonu olup olmadığını belirlemek amacıyla sadece kum kullanılarak yapılan ekstraksiyonda hi� DNA g�zlenmemiştir. Kum y�ntemi ve lizostafin ile yapılan ekstraksiyonlarda elde edilen DNA'ların saflıkları spektrofotometre ile �l��ld�kten sonra, 260/280 oranı iki y�ntemde de benzer şekilde 1.80 ve �zeri olarak belirlenmiştir. Kum y�ntemi ile elde edilen DNA'da PCR y�ntemini engelleyebilecek herhangi bir inhibit�r�n olup olmadığını araştırmak amacıyla, stafilokinaz ve stafilokok enterotoksin genlerine �zg�l primerler kullanılarak aynı koşullarda PCR yapılmış; bu primerlere �zg� bantlar elde edilmiştir. Cam boncuk kullanılarak elde edilen DNA'dan yapılan PCR sonucunda ise PCR �r�n�ne rastlanmamıştır (Resim 3).
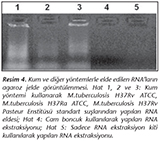
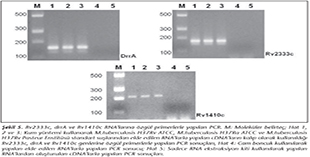
Standart �� mikobakteri suşuna kum y�ntemi uygulandığında, t�m�nden RNA elde edildiği halde, cam boncuk ve sadece RNA ekstraksiyon kiti kullanıldığında RNA elde edilmemiştir (Resim 4). Elde edilen RNA'nın kullanılabilirliğini araştırmak amacıyla cDNA sentezi ger�ekleştirilmiştir. Bu DNA'lar kullanılarak mikobakterilerin efluks pompası ile ilgili genlerinden olan ve daha �nceki �alışmamızda15 kullanmış olduğumuz Rv1410c, Rv2333c ve DrrA genlerine �zg�l primerlerle yapılan PCR sonucunda beklenen b�y�kl�kte PCR �r�nleri elde edilmiştir (Resim 5).
TARTIŞMA
Gram-pozitif bakterilerden etkin olarak DNA elde edilebilmesi i�in, h�cre duvarının lizostafin gibi enzimlerle veya mekanik par�alama y�ntemleriyle par�alanması gerekmektedir9. Bu �alışmada, mekanik olarak h�cre duvarının par�alanmasına dayanan kum y�nteminin etkinliği, lizostafin ve cam boncuk y�ntemleriyle kıyaslanmıştır. Kum y�ntemiyle, stafilokoklardan yapılan DNA ekstraksiyonundan elde edilen DNA'nın, lizostafin kullanıldığında elde edilen DNA miktarı ile aynı olduğu saptanmıştır. Yapılan spektrofotometrik analizler sonucunda, her iki y�ntemde elde edilen DNA'ların benzer saflıkta olduğu tespit edilmiştir. Kum y�nteminde kullanılan bakteri miktarı ve vorteksleme s�resi aynı olduğu halde, cam boncuk kullanılarak yapılan DNA ekstraksiyonunda, belirgin bir DNA eldesi olmamıştır. Bu sonucun, mekanik par�alama amacıyla kullanılan kumun y�zeyinin, cam boncuk y�zeyine g�re daha sert ve keskin olmasından kaynaklandığı d�ş�n�lm�şt�r. Gerek kum y�ntemi gerekse lizostafin y�ntemiyle elde edilen DNA'ların kalıp olarak kullanıldığı, bakteri kromozomunda farklı gen b�lgelerine �zg�l primerlerle yapılan PCR �alışmalarıyla, hedeflenen gen b�lgelerinin etkin olarak �oğaltıldığı g�zlenmiştir. DNA eldesine g�re �ok daha zor olan RNA ekstraksiyon işlemi amacıyla da kum y�ntemi kullanılmış ve RNA'lar elde edilmiştir. Elde edilen RNA'lardan oluşturulan cDNA'ların kalıp olarak kullanılmasıyla yapılan PCR �alışmalarında da hedef gen b�lgeleri �oğaltılmıştır.
�alışmamızda bakteri h�cre duvarının ortadan kaldırılması amacıyla geliştirilen kum y�ntemi, her ne kadar lizostafin ve cam boncuğun kullanıldığı y�ntemlerle kıyaslansa da, bu uygulamaları takip eden proteinaz K, fenol-kloroform ve etanol presipitasyon işlemleri ortaktır. �alışmamızda sunmamamıza karşın, DNA ekstraksiyonunda kullanılan diğer bir y�ntem olan Guanidin-silika y�ntemi, h�cre duvarı par�alandıktan sonra denenmiş ve proteinaz K ekstraksiyonu kadar DNA elde edilmiştir. Guanidin-silika y�ntemi, gerek gram-negatif gerekse memeli h�crelerinden direkt DNA elde edilmesinde etkili bir y�ntem olmasına karşın, kum y�ntemi kullanmadığımızda stafilokok DNA ekstraksiyonunda �alışmamıştır (veriler g�sterilmedi)16,17.
Bakteri h�cre duvarını par�alamak amacıyla silika ve zirkonia partik�llerinin kullanıldığı �alışmalar bulunmaktadır18. Başta silika olmak �zere her iki partik�l de DNA'ya y�ksek afinite ile bağlanmaktadır. Dolayısıyla, her ne kadar bu �alışmada test etmememize rağmen, bu iki molek�l�n kullanıldığı ekstraksiyon işlemleri sonucunda daha az miktarda DNA elde edilmesi ihtimali y�ksektir. Literat�r ve partik�llerin tanıtım bilgilerinde, bu partik�llerin DNA elde etmek amacıyla kullanılmadan �nce farklı kimyasallarla muamele edilmelerinin gerekliliği belirtilmektedir18. �alışmada kullandığımız kumun bulunması, hazırlanması, kullanılması ve bir kez elde edildikten sonra s�resiz olarak saklanması, diğer kimyasallara g�re avantaj sağlamaktadır. Bir diğer �nemli avantajı ise maliyetinin sıfır olmasıdır. Sonu� olarak, geliştirdiğimiz "Kum Y�ntemi" ile n�kleotid eldesi zor olan sert h�cre duvarına sahip bakterilerden saf ve yeterli miktarda DNA ve RNA elde edildiği g�zlenmiştir. Ayrıca pahalı sayılacak lizostafin veya farklı kimyasalların yerine, herhangi bir masrafı olmayan kum kullanılarak ekstraksiyon maliyeti d�ş�r�lebilmekte, DNA ekstraksiyon s�resi kısalmaktadır.
KAYNAKLAR
- Palomino JC. Molecular detection, identification and drug resistance detection in Mycobacterium tuberculosis. FEMS Immunol Med Microbiol 2009; 56(2): 103-11.
- Liu YT. A technological update of molecular diagnostics for infectious diseases. Infect Disord Drug Targets 2008; 8(3): 183-8.
- Caliendo AM. Multiplex PCR and emerging technologies for the detection of respiratory pathogens. Clin Infect Dis 2011; 52(4): 326-30.
- Lisby G. Application of nucleic acid amplification in clinical microbiology. Mol Biotechnol 1999; 12(1): 75-99.
- Bollet C, Gevaudan MJ, de Lamballerie X, Zandotti C, de Micco P. A simple method for the isolation of chromosomal DNA from gram positive or acid-fast bacteria. Nucleic Acids Res 1991; 19(8): 1955.
- Bergallo M, Costa C, Gribaudo G, et al. Evaluation of six methods for extraction and purification of viral DNA from urine and serum samples. New Microbiol 2006; 29(2): 111-9.
- Aldous WK, Pounder JI, Cloud JL, et al. Comparison of six methods of extracting Mycobacterium tuberculosis DNA from processed sputum for testing by quantitative real-time PCR. J Clin Microbiol 2005; 43(5): 2471-3.
- Thatcher SA. DNA/RNA preparation for molecular detection. Clin Chem 2015; 61(1): 89-99.
- Oliveira CF, Paim TG, Reiter KC, et al. Evaluation of four different DNA extraction methods in coagulase-negative staphylococci clinical isolates. Rev Inst Med Trop Sao Paulo 2014; 56(1): 29-33.
- Zhao J, Carmody LA, Kalikin LM, et al. Impact of enhanced Staphylococcus DNA extraction on microbial community measures in cystic fibrosis sputum. PLoS One 2012; 7(3): e33127.
- Amaro A, Duarte E, Amado A, et al. Comparison of three DNA extraction methods for Mycobacterium bovis, Mycobacterium tuberculosis and Mycobacterium avium subsp. avium. Lett Appl Microbiol 2008; 47(1): 8-11.
- Radomski N, Kreitmann L, McIntosh F, et al. The critical role of DNA extraction for detection of mycobacteria in tissues. PLoS One 2013; 8(10): e78749.
- Tan SC, Yiap BC. DNA, RNA, and protein extraction: the past and the present. J Biomed Biotechnol 2009; 2009: 574398.
- Sahin F, Karasartova D, Ozsan TM, et al. Identification of methicillin-resistant Staphylococcus aureus carrying an exfoliative toxin A gene encoding phage isolated from a hospitalized patient in Turkey. Can J Microbiol 2013; 59(4): 260-5.
- Calgin MK, Sahin F, Turegun B, et al. Expression analysis of efflux pump genes among drug-susceptible and multidrug-resistant Mycobacterium tuberculosis clinical isolates and reference strains. Diagn Microbiol Infect Dis 2013; 76(3): 291-7.
- Boom R, Sol CJ, Salimans MM, et al. Rapid and simple method for purification of nucleic acids. J Clin Microbiol 1990; 28(3): 495-503.
- Vingataramin L, Frost EH. A single protocol for extraction of gDNA from bacteria and yeast. Biotechniques 2015; 58(3): 120-5.
- Yuan S, Cohen DB, Ravel J, et al. Evaluation of methods for the extraction and purification of DNA from the human microbiome. PLoS One 2012; 7(3): e33865.
İletişim (Correspondence):
Prof. Dr. Fikret Şahin,
Ankara �niversitesi Tıp Fak�ltesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
Sıhhiye 06100, Ankara, T�rkiye.
Tel (Phone): +90 312 595 8277,
E-posta (E-mail): fsahin29@hotmail.com