İnvazif
Mantar Enfeksiyonlarının Tanımlanmasında Moleküler Yöntemlerin
Öneminin Konvansiyonel Yöntemler ile Karşılaştırılarak Değerlendirilmesi*
Evaluation
of the Significance of Molecular Methods in the Diagnosis of Invasive Fungal
Infections:
Comparison with Conventional Methods
Serdar SUSEVER, Yıldız YEĞENOĞLU
İstanbul Üniversitesi İstanbul Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, İstanbul.
Istanbul University Istanbul Faculty of Medicine, Department of Medical Microbiology, Istanbul, Turkey.
* Bu çalışma, İstanbul Üniversitesi Bilimsel Araştırma Projeleri Birimi tarafından desteklenmiştir (no:T329/03112003).
ÖZET
Özellikle riskli hasta gruplarında gerçek ya da fırsatçı patojen mantarların neden olduğu enfeksiyonların tanısında direkt mikroskobik inceleme ve kültür, halen değerini koruyan standart konvansiyonel yöntemlerdir. Bu yöntemlerin uygulama ve değerlendirmelerinde karşılaşılan bazı sorunlar nedeniyle, günümüzde daha hızlı, kolay uygulanabilir, yüksek duyarlılık ve özgüllüğe sahip yeni tanı yöntemlerine gereksinim duyulmaktadır. Bu çalışmada, klinik örneklerde etken olan mantarların tespiti ve tanımlanmasında moleküler yöntemler ile alınan sonuçların, eş zamanlı olarak uygulanan konvansiyonel tanı yöntemleri ile karşılaştırılması amaçlanmıştır. Çalışmaya, immünsüpresif 50 hastaya ait klinik örnekler [24 bronkoalveoler lavaj (BAL); 14 kan; beş periton, dört plevra ve bir perikard sıvısı; bir beyin omurilik sıvısı (BOS), bir idrar] dahil edilmiştir. Tüm örnekler sabouraud dekstroz ve beyin kalp infüzyon agar besiyerlerine ekilerek 25-30°C ve 37°Cde 30 gün boyunca inkübe edilmiş; kan dışındaki örnekler ise %10-15 KOH + kalkoflor beyazı ile boyanarak direkt mikroskopi ile incelenmiştir. İzolatların konvansiyonel olarak tanımlanmasında temel morfolojik ve biyokimyasal özellikler esas alınmıştır. Polimeraz zincir reaksiyonu (PCR) için örneklerden mantar DNAsının saflaştırılmasında, hem fenol-kloroform-izoamil alkol (9-10 saat süreli) hem de ticari DNA ekstraksiyon kiti (6-7 saat süreli) kullanılmıştır. PCR yöntemi, mantarların ITS1, ITS2, ITS3, ITS4, 5.8S rDNA ve 28S rDNA bölgelerinden seçilen genel ve türe özgül primerler (multipleks) kullanılarak uygulanmıştır. Genel primerler ile 550 baz çifti (bp) büyüklüğünde mantarlara özgül bantlar; türe özgül primerler ile de 273 bpde Candida albicans, 320 bpde Candida parapsilosis, 423 bpde Candida glabrata, 357 bpde Candida tropicalis, 385 bpde Aspergillus fumigatus ve 136 bpde Cryptococcus neoformansa özgül bantlar değerlendirilmiştir. Çalışılan 50 örneğin 17 (%34)sinin kültüründe üreme saptanmış (12 BAL, üç kan ve bir idrar örneğinde C.albicans, bir kan örneğinde C.parapsilosis), kan dışı 36 örneğin 7 (%19.4)si direkt mikroskopi ile pozitif bulunmuş (tümü BAL örneği) ve 27 (%54) örnek PCR ile olumlu sonuç vermiştir. Kültürlerinde üreme olan 17 örneğin tümünde PCR ile de pozitif sonuç alınmış, kültürü negatif 23 örnek ise PCR ile de negatif bulunmuştur. Direkt misroskopi ve kültürü negatif bulunan beş BAL, dört periton sıvısı ve bir BOS olmak üzere toplam 10 örnekte PCR ile mantar DNAları saptanmış ve multipleks PCR ile bunların sekizi C.albicans, biri C.parapsilosis (periton sıvısı örneği) ve biri de C.neoformans (BOS örneği) olarak tanımlanmıştır. PCR negatif, kültür pozitif bir sonuç elde edilmemiştir. Kültür negatif, PCR pozitif bulunan bu 10 örneğin antifungal tedavi alan hastalara ait olduğu izlenmiş, kriptokokoz şüpheli bir hastanın BOS örneğinde sadece PCR ile C.neoformans DNAsının saptanması, bu olguda hızla uygun tedaviye başlanmasına olanak sağlamıştır. İstatistiksel değerlendirmede, kültür ile PCR arasındaki uyum önemli düzeyde (k= 0.61; p< 0.001), mikroskopi ile PCR arasındaki uyum ise minimal düzeyde (k= 0.24; p< 0.001) saptanmış; kültür referans yöntem olarak alındığında, PCR yönteminin duyarlılığı %100, özgüllüğü ise %69.7 olarak belirlenmiştir. Çalışmamızda, kültürde üretilen ve konvansiyonel yöntemlerle tanımlanan tüm mantar türlerinin, uygulanan multipleks PCR ile de aynı şekilde tanımlandığı gözlenmiştir. Sonuç olarak, konvansiyonel yöntemler göz ardı edilmeksizin, hastanın klinik durumu ve laboratuvar koşulları dikkate alınarak, invazif mantar enfeksiyonlarının tanısında duyarlılığı yüksek bulunan PCR temelli testlerin kullanılmasının uygun olacağı düşünülmüştür.
Anahtar sözcükler: İnvazif mantar; konvansiyonel yöntem; polimeraz zincir reaksiyonu; multipleks PCR; Candida türleri; Cryptococcus neoformans; Aspergillus fumigatus.
ABSTRACT
Direct microscopy and culture methods are still valuable standard conventional methods for the diagnosis of infections caused by true or opportunistic fungal pathogens, especially in high risk patients. However, some of the problems concerning the application and interpretation of those methods, indicate a need for more rapid, practical and reliable tests with high sensitivity and specificity. This study was conducted to compare the results obtained by molecular methods with the results of conventional methods performed simultaneously for the detection and identification of causative fungi in clinical samples. Clinical samples [24 bronchoalveolar lavage (BAL); 14 blood; 5 peritoneal, 4 pleural and 1 pericardial fluids; 1 cerebrospinal fluid (CSF), 1 urine] from 50 immunosuppressed patients were included in the study. All of the samples were cultivated on Sabouraud dextrose and brain-heart infusion agar media and incubated at 30°C and 37°C for 30 days. Samples other than blood were stained with 10-15% KOH + calcofluor white and examined by direct microscopy. Conventional identification of the isolates were performed by using basic morphological and biochemical characteristics. The isolation of fungal DNAs for polymerase chain reaction (PCR) was achieved by classical phenol-chloroform-isoamylalcohol procedure (9-10 hours) and commercial DNA extraction kit (6-7 hours) and general and species-specific primers (multiplex) from ITS1, ITS2, ITS3, ITS4, 5.8S rDNA and 28S rDNA regions were choosen for amplification. In PCR results, 550 base-paired (bp) bands obtained with universal primers were evaluated as fungal DNA positivity, and 273 bp, 320 bp, 423 bp, 357 bp, 136 bp and 385 bp bands with species-specific primers were evaluated as Candida albicans, Candida parapsilosis, Candida glabrata, Candida tropicalis, Cryptococcus neoformans and Aspergillus fumigatus positivities, respectively. Seventeen (34%) of the 50 samples yielded fungal growth on culture (C.albicans in 12 BAL, 3 blood, 1 urine sample, and C.parapsilosis in 1 urine), while seven BAL out of 36 (19.4%) non-blood samples gave positive result by direct microscopy. Of the samples 27 (54%) were found positive by PCR. All of the 17 culture positive samples were also found PCR positive, and all of the 23 culture negative samples were also found PCR negative. However, fungal DNAs were detected by PCR in 10 of the samples (5 BAL, 4 peritoneal fluids, 1 CSF) which were negative by direct microscopy and culture methods. These fungi were identified as C.albicans (n= 8), C.parapsilosis (n= 1, from peritonal fluid) and C.neoformans (n= 1, from CSF) by multiplex PCR. No samples yielded PCR negative, culture positive result. All of those 10 PCR positive, culture negative samples belonged to patients who were under antifungal treatment. The detection of C.neoformans DNA from CSF sample of a patient with suspected cryptococcosis only with PCR provided the chance for rapid therapy. In statistical evaluation, the concordance between culture and PCR methods were found significantly high (k= 0.61; p< 0.001), whereas it was minimal (k= 0.24; p< 0.001) between direct microscopy and PCR. When considering culture as the reference method, the sensitivity and specificity of PCR were estimated as 100% and 69.7%, respectively. In addition, multiplex PCR was as successful as culture and conventional identification methods in the identification of all fungal species. As a result, without disregarding conventional methods, use of PCR might be recommended for the identification of fungal species on the basis of clinical status of the patient and conditions of the laboratory.
Key words: Invasive fungi; conventional method; polymerase chain reaction; multiplex PCR; Candida species; Cryptococcus neoformans; Aspergillus fumigatus.
Geliş Tarihi (Received): 08.10.2010 • Kabul Ediliş Tarihi (Accepted): 24.11.2010
GİRİŞ
Son yıllarda mantar enfeksiyonlarının öneminin artmasından dolayı etken olan mantarların tanısı, tür tayini, tiplendirilmesi ve antifungal ilaç direnci ile ilgili çalışmalar hız kazanmıştır. Özellikle immün sistemi baskılanmış hastalarda erken tanı ve tedavi, yüksek mortalite hızını düşürmek açısından çok önemlidir. İnvazif mantar hastalıklarının tanımlanmasında direkt mikroskobik inceleme ve kültür, standart konvansiyonel yöntemlerdir. Ancak gerek zaman alıcı olması, gerekse her zaman olumlu sonuç vermemeleri ve bazı mantarlar açısından zor uygulanabilir ve yorumlanabilir olmaları nedeniyle, bu yöntemlerin yanı sıra hızlı, duyarlılık ve özgüllüğü yüksek yeni tanı yöntemleri geliştirilmektedir. Son yıllarda moleküler yöntemlerin giderek artan oranlarda uygulanması, bu güçlüklerin çözümlenmesinde önemli bir adımdır1,2,3,4,5.
Mantar enfeksiyonlarının moleküler tanısı, ya doğrudan hasta örneği ya da kültür ortamından gerçekleştirilebilir. Moleküler tanının ilk basamağında öncelikle, hastada varlığından kuşku duyulan mantar hastalığının kanıtlanması, ikinci aşamada ise enfeksiyon etkeni olan mantarın tür düzeyinde tanımlanması amaçlanmıştır. Mantarların tanımlanmasında en yaygın kullanılan iki DNA bölgesi, küçük ve büyük ribozomal alt üniteleri arasında yer alan türe özgül internal transcribed spacer (ITS) ve büyük ribozomal alt ünitesinin (26S) D1/D2 bölgeleridir. Bu bölgeler tür düzeyinde farklılıklar gösteren yeterli zincir heterojenitesi içermektedir. Dolayısıyla mantarları tür düzeyinde tanımlamak ve olası mutasyonel değişiklikleri saptamak için daha uygundur. Mantarların genotiplendirme çalışmalarında, protein kodlayan intron bölgeler ve ribozomal DNA (rDNA) gibi protein kodlamayan gen bölgeleri hedef olarak kullanılır2,3,4,5. ITSnin yanı sıra en çok tercih edilen genomik alanlar, mitokondriyal sitokrom b, filogenetik yönden bilgi sağlayan genetik moleküller (large subunit rDNA), P450L1A1 dimetilaz ve aktin bölgeleridir.
Polimeraz zincir reaksiyonu (PCR) yönteminde, birden fazla türe özgül primer çiftinin kullanılması sonucunda multipleks PCR testi geliştirilmiştir. Bu teknikte, ilk aşamada geniş özgüllüğü olan DNA ürünü (örn. ITS bölgesi) çoğaltılır ve daha sonra bu ürün türe özgül primerler ile reaksiyona sokulup, DNA ürünlerinin jel üzerinde farklı bantlar oluşturması izlenir6. Diğer mikroorganizmalara bağlı yalancı pozitifliği ortadan kaldıran bu yöntem ile sekiz saat gibi kısa bir sürede ve aynı reaksiyon boyunca birden fazla mantarın tür düzeyinde tanısını yapabilmek mümkün olmaktadır7.
Bu çalışmada, klinik örneklerde etken olan mantarların moleküler yöntemler ile tespiti ve tür düzeyinde tanımlanmaları ve sonuçların eş zamanlı olarak uygulanan konvansiyonel tanı yöntemleriyle karşılaştırılması amaçlanmıştır.
GEREÇ ve YÖNTEM
Çalışmaya, İstanbul Üniversitesi İstanbul Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Mikoloji Bilim Dalına gönderilen, immünsüpresif hastalara ait 24 bronkoalveoler lavaj (BAL) sıvısı, 14 kan, beş periton diyaliz sıvısı, dört plevra sıvısı ve birer adet olmak üzere beyin omurilik sıvısı (BOS), perikard sıvısı ve idrar örneği dahil edildi. Referans suş olarak, İstanbul Tıp Fakültesi Mikroorganizma ve Kültür Koleksiyonları Merkezi (KÜKENS)nden sağlanan Candida albicans ATCC 10231, Candida parapsilosis ATCC 22019, Candida glabrata ATCC 15126, Candida tropicalis KÜEN 1025, Cryptococcus neoformans ATCC 90112 ve Aspergillus fumigatus ATCC 9197 suşları kullanıldı.
Sistemik mikoz ön tanılı hastalardan alınan klinik örneklere %10-15 KOH + kalkoflor beyazı eklenerek direkt preparatlar hazırlandı ve mantar elemanları yönünden floresan mikroskopla incelendi. Aynı zamanda her örnek sabouraud dekstroz agar (SDA) (pH= 5.5-6) ve beyin kalp infüzyon agar (BHIA) besiyerlerine ekilerek 25-30°C ve 37°C olmak üzere iki farklı sıcaklıkta 30 gün boyunca inkübe edildi. İzole edilen mayaların tanımlanmasında koloni görünümü ve rengi, hif ve/veya yalancı hif oluşumu, klamidospor, artrokonidya, blastokonidya oluşturmaları, germ tüp oluşturma yetenekleri, kapsül varlığı, karbonhidrat, nitrat asimilasyonları ve şekerleri fermente edebilme gibi temel morfolojik ve biyokimyasal özellikler esas olarak alındı8.
Örneklerden DNA izolasyonu, ticari bir ekstraksiyon kiti ve fenol-kloroform-izoamil alkol yöntemi7 ile yapıldıktan sonra, mantarların tür düzeyinde tanımlanması için multipleks PCR, özgül primerler kullanılarak Luo ve arkadaşlarının9 bildirdiği yöntemin modifikasyonu ile uygulandı. PCR karışımı, toplam hacim 20 µl olacak şekilde; 2 µl (~1 ng) DNA, 20 mM tris-HCl (pH: 8.4), 50 mM KCl, dNTP (her biri için 0.2 mM, dATP, dCTP, dGTP, dTTP), 0.5 µmol primer ve 0.5 U Taq polimeraz ile hazırlandı. PCR döngüsü; 96°Cde 5 dakika (1 döngü), 94°Cde 30 saniye, 58°Cde 30 saniye, 72°Cde 30 saniye (40 döngü) ve 72°Cde 15 dakika (1 döngü) olarak gerçekleştirildi. Elde edilen PCR ürünleri %1lik agaroz jelde elektroforetik olarak 100 volt altında yaklaşık 30 dakika yürütüldü ve bantlar ultraviyole altında görüntülendi.
Genel primerler olarak U1: GTGAAATTGTTGAAAGGGAA ve U2: GACTCCTTGGTCCGTGTT (~550 baz çifti; bp), türe özgül primerler olarak da A.fumigatus için AFUM1: CGCCGAAGACCCCAACAT GAACGC ve AFUM2: TAAAGTTGGGTGTCGGCTGGC (GenBank AF176 62, AF078889) (~385 bp), C.albicans için CALB1: TTTATCAACTTGTCACACCAGA ve CALB2: ATCCCGCCTTACCACTACCG (GenBank 47111, L28817) (~273 bp), C.glabrata için CGL1: TTATCACACGACTCGACACT ve CGL2: CCCACATACTGATATGGCCTACAA (GenBank AB032177, AF167993) (~ 423 bp), C.parapsilosis için CPA1: TTGGTAGGCCTTCTATATGGG ve CPA2: GCCAGAGATTAAACTCAACCAA (GenBank AF287909, L47109) (~320 bp), C.tropicalis için CTR1:CAATCCTACCGCCAGAGGTTAT ve CTR2: TGGCCACTAGCAAAATAAGCGT (GenBank AF287910, AF268095) (~357 bp), C.neoformans için CN4: GAAGGGCATGCCTGTTTGAGAG ve CN5: ATCACCTTCCCACTAACACATT (GenBank M94516, M64517) (~136 bp) seçildi. Bu yöntemde modifikasyonlar yapılarak altı mantar türü için iki multipleks PCR paneli (TP ve TAN paneli) test edildi. TP panelindeki primerler; C.tropicalis (CTR1, CTR2) ve C.parapsilosisin (CPA1, CPA2), TAN panelindeki primerler, C.tropicalis (CTR1, CTR2), C.albicans (CALB1, CALB2) ve C.neoformansın (CN4, CN5) tanımlanmasında kullanıldı.
İstatistiksel değerlendirmede, iki veya daha fazla veri arasındaki kalitatif uyum oranını (k: Kappa) ölçmek için geliştirilen Cohen Kappa analiz testi kullanıldı. Yöntemler arasında belirlenen k değerinin 0 olması şansa bağlı uyum, < 0.20 olması önemsiz uyum, 0.21-0.40 olması minimal uyum, 0.41-0.60 olması orta derecede uyum, 0.61-0.80 olması önemli derecede uyum ve 0.81-1.00 olması tam uyum olarak değerlendirildi10.
BULGULAR
Çalışmaya alınan 50 klinik örnek, eş zamanlı olarak moleküler (PCR) ve konvansiyonel (direkt mikroskopi ve kültür) yöntemleriyle incelenerek değerlendirilmiştir. Örneklerin 17 (%34)sinin kültüründe üreme saptanmış (16 C.albicans, bir C.parapsilosis), 33 (%66) örnekte ise üreme olmamıştır (Tablo I). Örneklerin 14ü kan olduğu için, direkt mikroskopi ve kültür uyumu 36 örnek üzerinden değerlendirilmiştir. Buna göre, 7 (%19.4) örneğin; hem mikroskopisi hem de kültürü pozitif bulunurken, 6 (%16.7) örneğin mikroskopisi negatif, kültürü pozitif olarak saptanmıştır. Yirmi üç (%63.8) örnekte ise her iki yöntemle de negatif sonuç alınmıştır. Direkt mikroskopisi pozitif, kültürü negatif bir örneğe rastlanmamıştır (Tablo I).
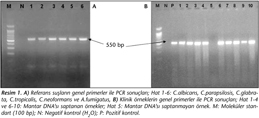
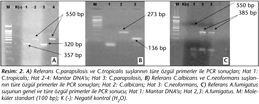
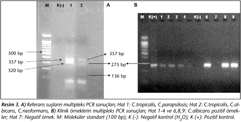
Genel primerler ile yapılan PCR sonucunda, örneklerin 27 (%54)sinde mantar DNAsı saptanmış; bu pozitif örneklerin dördü kan olduğu için direkt mikroskobik inceleme sadece 23 örnekte gerçekleştirilmiş ve yedisinde pozitif olarak bulunmuştur. PCR ile pozitif bulunan örneklerin 17sinin kültürü pozitif, 10unun ise negatif saptanmış; PCR negatif, kültür pozitif bir sonuç elde edilmemiştir. Kültürlerinde üreme olan 17 örneğin tümünde PCR ile de pozitif sonuç alınmış, kültürü negatif 23 örnek ise PCR ile de negatif sonuç vermiştir. Çalışmamızda, referans suşlar ve klinik örneklerden elde edilen bazı PCR sonuçları Resim 1, 2, 3te gösterilmiştir.
Mantar DNAsı pozitif bulunan 27 örneğe türe özgül multipleks PCR yöntemi uygulandığında; 24 (%88.8)ünün C.albicans, 2 (%7.4)sinin C.parapsilosis ve 1 (%3.7)inin C.neoformans olduğu belirlenmiştir (Tablo I). Kültür pozitifliği saptanan 12 BAL, dört kan ve bir idrar örneğinde konvansiyonel yöntemlerle tanımlanan Candida türlerinin, multipleks PCR ile de aynı şekilde tanımlandığı izlenmiştir. Buna karşın direkt mikroskopisi ve kültürü negatif bulunan beş BAL, dört periton sıvısı ve bir BOS olmak üzere toplam 10 örnekte PCR ile mantar DNAları saptanmış ve multipleks PCR ile bunların sekizi C.albicans, biri C.parapsilosis (periton sıvısı örneği) ve biri de C.neoformans (BOS örneği) olarak tanımlanmıştır (Tablo I).
Çalışmada kullanılan iki ekstraksiyon yöntemi karşılaştırıldığında aralarında anlamlı bir fark olmadığı görülmüş; sadece fenol-kloroform-izoamil alkol (FKİ) yönteminde bir BAL örneğinde, ticari kit ile elde edilemeyen mantar DNAsı saptanmıştır. Ticari kit ile DNAnın elde edilmesi 6-7 saatte tamamlanırken, bu süre FKİ yönteminde yaklaşık 9-10 saat olarak belirlenmiştir.
İstatistiksel analiz sonunda, kültür ile PCR arasındaki uyum önemli düzeyde (k= 0.61; p< 0.001), mikroskopi ile PCR arasındaki uyum ise minimal düzeyde (k= 0.24; p< 0.001) saptanmış; kültür referans yöntem olarak alındığında, PCR yönteminin duyarlılığı %100, özgüllüğü ise %69.7 olarak belirlenmiştir.
TARTIŞMA
Günümüzde mantarların tanımlanmasında kullanılan fenotipik yöntemlerde karşılaşılan sorunlar, moleküler yöntemlerin; mikrobiyolojinin diğer alanlarında olduğu gibi gerektiğinde mikoloji alanında da kullanılmasını zorunlu hale getirmiş, tanı, tedavi izlemi ve epidemiyoloji konusunda giderek başarılı sonuçlar alınmasını sağlamıştır. 1990lı yılların ikinci yarısından itibaren mantarların, özellikle de immün sistemi baskılanmış hastalar için büyük öneme sahip Aspergillus ve Candida türlerinin klinik örneklerde saptanması ile ilgili çalışmalar hız kazanmıştır11,12,13,14,15,16. Skladny ve arkadaşları17, 315 klinik örnekte yaptıkları çalışmada, Aspergillus türlerine özgül primerlerin kullanıldığı PCR yönteminin duyarlılığını %100, özgüllüğünü ise yaklaşık %89 olarak bildirmişlerdir. Ülkemizde Kalkancı ve arkadaşları18, nötropenisi olan 49 hastanın kan örneklerinde PCR ile mantar DNAsı araştırmışlar, 7 (%14.2)sinde genel (panfungal) primerlerle ve 5 (%10.2)inde Candida primerleriyle olumlu sonuç bildirmişlerdir.
Yapılan çalışmalarda, mantarların tanımlanmasına yönelik moleküler yöntemlerde ITS1-4, 5.8S rDNA ve 28S rDNA gibi farklı gen bölgelerini hedef alan primerlerin kullanıldığı görülmektedir19,20,21. Li ve arkadaşları22, 234 kan örneğinden izole ettikleri maya türleri (121 C.albicans, 50 C.tropicalis, 29 C.glabrata, 20 C.parapsilosis, altı C.neoformans ve diğer) ile 18S rDNA, ITS1, 5.8S rDNA, ITS2, ITS4 ve 26S rDNA gen bölgelerini hedefleyen primerler ile örneklere doğrudan uyguladıkları multipleks PCR sonucu tanımlanan maya türlerinin uyumlu olduğunu saptamışlar; multipleks PCRnin duyarlılığını %98.7 olarak bildirmişlerdir. Bir başka çalışmada ise, invazif aspergilloz şüpheli 10, kolonizasyon şüpheli beş ve aspergilloz şüphesi olmayan 162 olgudan alınan 177 BAL örneği, kültür ve PCR yöntemleriyle araştırılmış; PCR yönteminin duyarlılığı %100, özgüllüğü ise %96 olarak rapor edilmiştir23. Klinik örneklerde mantarların moleküler tanısı ile ilgili yapılan diğer çalışmalarda da, özgüllük oranları ile ilgili bazı farklı sonuçlara rağmen, PCR temelli yöntemlerin duyarlılıklarının konvansiyonel yöntemlere göre daha yüksek olduğu konusunda görüş birliği vardır9,12,15,16,17,18,24,25,26,27.
Sunulan bu çalışmada, immünsüpresif 50 hastaya ait klinik örneklerin 17 (%34)sinde kültür ve 27 (%54)sinde multipleks PCR yöntemiyle olumlu yanıt alınmış; 23 (%46) örnek ise tüm yöntemlerle negatif bulunmuştur (Tablo I). Kültür ile pozitif bulunan tüm örnekler PCR ile de pozitif sonuç vermiş; PCR ile pozitif saptanan 10 örneğin kültürlerinde üreme olmamıştır. Kültürü pozitif, PCR sonucu negatif örneğe ise rastlanmamıştır. Buna göre; kültür ile PCR arasında önemli derecede uyum (k= 0.61; p< 0.001) gözlenmiş ve PCR yönteminin duyarlılığı %100, özgüllüğü ise %69.7 olarak belirlenmiştir. Çalışmamızda saptanan yüksek PCR pozitiflik oranı, Yamakami ve arkadaşları13 ile Morace ve arkadaşlarının16 sonuçlarıyla benzerlik göstermektedir.
Maaroufi ve arkadaşları26, antifungal tedavi alan hastaların klinik örneklerinde kültür ile negatif, PCR ile pozitif sonuç aldıklarını bildirmişler ve antifungal tedavinin, mantarların kültürde üremesini baskılayabileceğini ifade etmişlerdir. Bizim çalışmamızda da, 10 örneğin kültüründe üreme olmamasına karşın PCR ile pozitif sonuç alınması, hastaların antifungal tedavi almalarından kaynaklanmış olabilir. Nitekim bu hastaların öyküsünde, örneklerin gönderildiği dönemde antifungal tedavi aldıkları belirlenmiş, ancak tedavi ile ilgili ayrıntılı klinik bilgilere ulaşılamamıştır.
Çalışmaya alınan klinik örneklerin kültürlerinden izole edilen 17 Candida suşunun 16sı C.albicans (12 BAL, üç kan ve bir idrar izolatı) olarak tanımlanmış, kan örneklerinin birinden ise C.parapsilosis üretilmiştir. Bu bulgu, C.albicansın fungal etkenler arasında halen ilk sırayı koruduğunu bildiren çalışmalarla uyumludur14,16,22,24. Çalışmamızda, konvansiyonel yöntemler ile pozitif sonuç alınamayan kriptokokoz şüpheli bir hastanın BOS örneğinde PCR ile C.neoformans DNAsının saptanması, bu olguda hızla uygun tedaviye başlanmasına olanak sağlamış ve moleküler tanının önemini bir kez daha vurgulamıştır.
Çalışmamızda, kültürde üretilen ve konvansiyonel yöntemlerle tanımlanan Candida türlerinin tamamının, uyguladığımız multipleks PCR yöntemiyle de aynı şekilde tanımlanmış olması, bu yöntemin, klinik örneklerde mantarların tür düzeyinde tanımlanmasında başarıyla kullanılabileceğini düşündürmüştür. Birçok araştırıcı, multipleks PCR ile yaptıkları çalışmalarda, çeşitli mantarlara ait 5.8S rDNA, 28S rDNA, 18S rDNA, 26S rDNA, ITS1, ITS2-ITS3 ve ITS4 gibi gen bölgelerini çoğaltan primerleri kullanmışlar ve tanımlamada farklı bant büyüklüklerini [C.albicans (273 bp, 279 bp, 386 bp, 402 bp, 218/219 bp ve 110 bp), C.glabrata (423 bp, 359 bp, 234 bp, 632 bp, 482/483 bp), C.parapsilosis (320-300 bp, 251 bp, 336 bp, 126 bp, 229 bp), C.tropicalis (357 bp, 268 bp, 373 bp, 149 bp, 218 bp), C.neoformans (136 bp, 316 bp, 516 bp, 201 bp), A.fumigatus (385 bp, 284 bp)] dikkate almışlardır9,19,20,22,25. Luo ve arkadaşları9, multipleks PCR protokolünü uygularken, altı mantar türünü A.fumigatus, C.albicans, C.neoformans ve C.glabrata, C.tropicalis, C.parapsilosis olarak gruplandırmış ve iki panel şeklinde çalışmışlardır. Çalışmamızda da önce bu araştırıcıların uyguladığı protokol kullanılmış, ancak çok sayıda yalancı bandın tespit edilmesi nedeniyle, bu protokol modifiye edilerek (1. panel: C.tropicalis, C.parapsilosis; 2. panel: C.tropicalis, C.albicans, C.neoformans) uygulanmış ve yalancı bant oluşumu engellenmiştir. Buna göre çalışmamızda, C.albicans için 273 bp, C.glabrata için 423 bp, C.parapsilosis için 320 bp, C.tropicalis için 357 bp, C.neoformans için 136 bp ve A.fumigatus için 385 bp büyüklüğündeki bantlar pozitif olarak değerlendirilmiştir (Resim 1, 2, 3).
Çalışmamızda mantar DNAlarının izolasyonu amacıyla, klasik FKİ yönteminin yanı sıra ticari bir ekstraksiyon kiti de kullanılmış ve yöntemler arasında önemli bir fark olmadığı görülmüştür. Ancak FKİ yönteminde, bir örnekte (BAL) ticari kit ile elde edilemeyen mantar DNAsı saptanmıştır. Löffler ve arkadaşlarının çalışmasında da, konvansiyonel DNA ekstraksiyon yöntemi ile ticari kit arasındaki uyum %99 olarak bildirilmektedir15.
Sonuç olarak, gerek klinik örneklerde mantar varlığının saptanmasında gerekse etkenlerin tür düzeyinde tanımlanmasında konvansiyonel yöntemlerle yüksek düzeyde uyum göstermesi ve yaklaşık %70 olarak belirlenen özgüllüğüne rağmen duyarlılığının %100 olarak saptanmış olması, PCR yönteminin hızlı tanıda kullanılabilecek bir seçenek olduğunu düşündürmektedir. Ancak yine de, daha geniş hasta gruplarının değerlendirildiği ileri çalışmalara gereksinim olduğu açıktır.
KAYNAKLAR
- Çerikçioğlu N. Yeni identifikasyon yöntemleri, s:182-90. Yeğenoğlu Y, Erturan Z (ed), 3.Ulusal Mantar Hastalıkları ve Klinik Mikoloji Kongresi, Kongre Kitabı. 27-30 Mayıs 2003, Bodrum. Türk Mikrobiyoloji Cemiyeti Yayınları No: 46. Çatı Grafik, İstanbul.
- Kurtzman CP. Molecular taxonomy of the yeasts. Yeast 1994; 10(13): 1727-40. [Özet]
- Saraçlı MA. Candida infeksiyonlarının laboratuvar tanısı: Moleküler ve genetik tanı yöntemleri, s: 133-44. Kiraz N, Kiremitçi A, Akgün Y (ed), Candida Mikrobiyolojisi ve İnfeksiyonları Simpozyumu, Tutanaklar. 21-22 Haziran 2002, Eskişehir. Türk Mikrobiyoloji Cemiyeti Yayınları No: 43. OGÜ Basımevi, Eskişehir.
- Saraçlı MA. Mikozların moleküler tanısı: Neredeyiz? s: 33-45. Tuncer İ, Fındık D, Arslan U, (ed), 4.Ulusal Mantar Hastalıkları ve Klinik Mikoloji Kongresi, Kongre Kitabı. 3-6 Mayıs 2005, Konya. Türk Mikrobiyoloji Cemiyeti Yayınları No: 49. Çatı Grafik, İstanbul.
- Valente P, Ramos JP, Leoncini O. Sequencing as a tool in yeast molecular taxonomy. Can J Microbiol 1999; 45(11): 949-58. [Özet]
- Zhao J, Kong F, Li R, Wang X, Wan Z, Wang D. Identification of Aspergillus fumigatus and related species by nested PCR targeting ribosomal DNA internal transcribed spacer regions. J Clin Microbiol 2001: 39(6): 2261-6. [Özet] [Tam Metin] [PDF]
- Kaufman PB, Wu W, Kim D, Cseke LJ. Handbook of Molecular and Cellular Methods in Biology and Medicine. 1995, CRC Press, Boca Raton, Florida.
- Kwon-Chung KJ, Bennet JE. Medical Mycology. 1992. Lea & Febiger, Pennsylvania.
- Luo G, Mitchell TG. Rapid identification of pathogenic fungi directly from cultures by using multiplex PCR. J Clin Microbiol 2002; 40(8): 2860-5. [Özet] [Tam Metin] [PDF]
- Landis JR, Koch GG. The measurement of observer agreement for categorical data. Biometrics 1977; 33(1): 159-74. [Özet]
- Spreadbury C, Holden D, Aufauvre-Brown A, Bainbridge B, Cohen J. Detection of Aspergillus fumigatus by polymerase chain reaction. J Clin Microbiol 1994; 32(8): 2039.
- Bretagne S, Costa JM, Marmorat-Khuong A, et al. Detection of Aspergillus species DNA in bronchoalveolar lavage samples by competitive PCR. J Clin Microbiol 1995; 33(5): 1164-8. [Özet] [PDF]
- Yamakami Y, Hashimoto A, Tokimatsu I, Nasu M. PCR detection of DNA specific for Aspergillus species in serum of patients with invasive aspergillosis. J Clin Microbiol 1996; 34(10): 2464-8. [Özet] [PDF]
- Einsele H, Hebart H, Roller G, et al. Detection and identification of fungal pathogens in blood by using molecular probes. J Clin Microbiol 1997; 35(6): 1353-60. [Özet] [PDF]
- Löffler J, Hebart H, Schumacher U, Reitze H, Einsele H. Comparison of different methods for extraction of DNA of fungal pathogens from cultures and blood. J Clin Microbiol 1997; 35(12): 3311-2. [Özet] [PDF]
- Morace G, Pagano L, Sanguinetti M, et al. PCR-restriction enzyme analysis for detection of Candida DNA in blood from febrile patients with hematological malignancies. J Clin Microbiol 1999; 37(6): 1871-5. [Özet] [Tam Metin] [PDF]
- Skladny H, Buchheidt D, Baust C, et al. Specific detection of Aspergillus species in blood and bronchoalveolar lavage samples of immunocompromised patients by two-step PCR. J Clin Microbiol 1999; 37(12): 3865-71. [Özet] [Tam Metin] [PDF]
- Kalkancı A, Kuştimur S, Güneş İ, Otlu B. Gazi Üniversitesi Tıp Fakültesi Mikrobiyoloji ve Klinik Mikrobiyoloji Anabilim Dalında invazif mantar infeksiyonlarının tanısında polimeraz zincir reaksiyonunun kullanılması, s: 178. Kiraz N, Kiremitçi A, Akgün Y (ed), Candida Mikrobiyolojisi ve İnfeksiyonları Simpozyumu, Tutanaklar. 21-22 Haziran 2002, Eskişehir. Türk Mikrobiyoloji Cemiyeti Yayınları No: 43. OGÜ Basımevi, Eskişehir.
- Turenne CY, Sanche SE, Hoban DJ, Karlowsky JA, Kabani AM. Rapid identification of fungi by using the ITS2 genetic region and an automated fluorescent capillary electrophoresis system. J Clin Microbiol 1999; 37(6): 1846-51. [Özet] [Tam Metin] [PDF]
- Bougnoux M, Dupont C, Mateo J, et al. Serum is more suitable than whole blood for diagnosis of systemic candidiasis by nested PCR. J Clin Microbiol 1999; 37(4): 925-30. [Özet] [Tam Metin] [PDF]
- Becker K, Badehorn D, Keller B, et al. Isolation and characterization of a species-specific DNA fragment for identification of Candida (Torulopsis) glabrata by PCR. J Clin Microbiol 2001; 39(9): 3356-9. [Özet] [Tam Metin] [PDF]
- Li YL, Leaw SN, Chen JH, Chang HC, Chang TC. Rapid identification of yeasts commonly found in positive blood cultures by amplification of the internal transcribed spacer regions 1 and 2. Eur J Clin Microbiol Infect Dis 2003; 22(11): 693-6. [Özet]
- Hayette MP, Vaira D, Susin F, et al. Detection of Aspergillus species DNA by PCR in bronchoalveolar lavage fluid. J Clin Microbiol 2001; 39(6): 2338-40. [Özet] [Tam Metin] [PDF]
- Fujita SI, Senda Y, Nakaguchi S, Hashimoto T. Multiplex PCR using internal transcribed spacer 1 and 2 regions for rapid detection and identification of yeast strains. J Clin Microbiol 2001; 39(10): 3617-22. [Özet] [Tam Metin] [PDF]
- Chang HC, Leaw SN, Huang AH, Wu TL, Chang TC. Rapid identification of yeasts in positive blood cultures by a multiplex PCR method. J Clin Microbiol 2001; 39(10): 3466-71. [Özet] [Tam Metin] [PDF]
- Maaroufi Y, Heymans C, De Bruyne JM, et al. Rapid detection of Candida albicans in clinical blood samples by using a TaqMan-based PCR assay. J Clin Microbiol 2003; 41(7): 3293-8. [Özet] [Tam Metin] [PDF]
- Khot PD, Fredricks DN. PCR-based diagnosis of human fungal infections. Expert Rev Anti Infect Ther 2009; 7(10): 1201-21. [Özet]
İletişim (Correspondence):
Dr. Serdar Susever,
İstanbul Üniversitesi İstanbul Tıp Fakültesi,
Tıbbi Mikrobiyoloji Anabilim Dalı,
Mikoloji Bilim Dalı,
Çapa, İstanbul, Türkiye.
Tel (Phone): +90 212 414 2000/32602,
E-posta (E-mail): ssusever2@yahoo.com