Viridans Grup Streptokok Tanımlamasında MALDI-TOF ve 16S rRNA Y�ntemlerinin Karşılaştırılması
Comparison of MALDI-TOF and 16S rRNA Methods in Identification of Viridans Group Streptococci
Serap S�Z�K YILDIZ1, Banu KAŞKATEPE2, Salih ALTINOK1, Mustafa �ETİN3, Alper KARAG�Z1, S�meyra SAVAŞ4
1 T�rkiye Halk Sağlığı Kurumu, Mikrobiyoloji Referans Laboratuvarları Daire Başkanlığı, Ankara.
1 Public Health Agency of Turkey, Department of Microbiology Reference Laboratories, Ankara, Turkey.
2 Ankara �niversitesi, Eczacılık Fak�ltesi, Farmas�tik Mikrobiyoloji Anabilim Dalı, Ankara.
2 Ankara University, Faculty of Pharmacy, Department of Pharmaceutical Microbiology, Ankara, Turkey.
3 Ankara Numune Eğitim ve Araştırma Hastanesi, Kardiyoloji Kliniği, Ankara.
3 Ankara Numune Training and Research Hospital, Department of Cardiology, Ankara, Turkey.
4 T�BİTAK BİLGEM Hesaplamalı Biyoloji İleri Genom ve Biyoenformatik Araştırma Merkezi, Kocaeli.
4 TUBITAK IGBAM Advanced Genomics and Bioinformatic Research Center, Kocaeli, Turkey.
�Z
Enfektif endokardit etkeni olarak sıklıkla karşımıza �ıkan viridans grup streptokoklar (VGS)'ın doğru tanımlanması her zaman klinik mikrobiyoloji laboratuvarı i�in sorun oluşturmaktadır. Klinik mikrobiyoloji laboratuvarları genellikle bu bakterilerin tanımlamasında konvansiyonel y�ntemler ile birlikte yarı otomatize/otomatize sistemler ve molek�ler y�ntemleri kullanmaktadır. Son zamanlarda VGS tanısında "Matriks Yardımlı Lazer İyonizasyon K�tle Spektrometre, Matriks Assisted Laser Ionization-Time of Flight" MALDI-TOF sistemlerinin kullanıldığı �alışmalar yayımlanmaktadır. Bu �alışmada, enfektif endokardit a�ısından riskli kişilerin ağız mikrobiyotasındaki VGS'lerin tanısında kullanılan konvansiyonel y�ntemler, yarı otomatize ve MALDI-TOF MS sistemlerinin altın standart y�ntem 16S rRNA dizi analizi ile karşılaştırarak performanslarının belirlenmesi ve elde edilen veriler doğrultusunda klinik mikrobiyoloji laboratuvarında VGS identifikasyonu i�in kullanılabilecek bir tanı algoritmasının oluşturulması ama�lanmıştır. �alışmaya Şubat-Haziran 2015 tarihleri arasında Ankara Numune Eğitim ve Araştırma Hastanesi, Kardiyoloji Polikliniğine başvuran enfektif endokardit gelişme riski bulunan romatizmal kalp hastalığı, kapak hastalığı ve/veya prostetik kapağı olan hastaların ağız mikrobiyotasından elde edilen 51 VGS izolatı dahil edilmiştir. Bakterilerin izolasyonu i�in standart mikrobiyolojik y�ntemler ile optokin ve safrada erime testi uygulanmıştır. Bakteriler API STREP (bioM�rieux, Fransa) ve MALDI-TOF MS Bruker Microflex (Bruker Biotyper; Bruker Daltonics, Bremen, Almanya) y�ntemleri ile tanımlanmıştır. Bakterilerin 16S rRNA dizi analizinde BSF-8 (5�-AGAGTTTGATCCTGGCTCAG-3�) ve BSR-534 (5�-ATTACCGCGGCTGCTGGC-3�) primerleri kullanılmıştır. ABI PRISM 3100 Avant Genetic Analyzer (Applied Biosystems, Foster City, CA, ABD) cihazı kullanılarak dizi analizi ger�ekleştirilmiştir. Elde edilen elektroferogramlar, Seq Scape Software (Applied Biosystems, Foster City, CA, ABD) programı ile analiz edilerek BLASTN (NCBI) programı ile GenBank referans dizileri ile karşılaştırılmıştır. Optokin ve safrada erime testleri sonucunda optokin ve safraya diren�li alfa hemolitik streptokokların API STREP sistemi ile 16 (%31.37)'sının Mitis grubunda, 15 (%29.41)'inin Anginosus grubunda, 9 (%17.65)'unun Salivarius grubunda, 7 (%13.73)'sinin Sanguinis grubunda ve 4 (%7.84)'�n�n Bovis grubunda yer aldığı tespit edilmiştir. MALDI-TOF sistemi ile aynı izolatların 20 (%39.22)'sinin Mitis grubunda, 14 (%27.45)'�n�n Anginosus grubunda, 13 (%25.49)'�n�n Salivarius grubunda ve 4 (%7.84)'�n�n Sanguinis grubunda yer aldığı tanımlanmıştır. 16S rRNA ile yapılan tanımlamada ise izolatların 25 (%49.02)'inin Mitis grubunda, 13 (%25.49)'�n�n Anginosus grubunda, 12 (%23.53)'sinin Salivarius grubunda ve 1 (%1.96)'inin Sanguinis grubunda yer aldığı bulunmuştur. Elde edilen sonu�lara g�re MALDI-TOF MS sisteminde tanımlanan 33 (%64.70) izolatın, API STREP sisteminde tanımlanan 31 (%60.78) izolatın 16s rRNA dizi analizi y�ntemi ile uyumlu olduğu bulunmuştur. Mitis grubu i�in API STREP testinin duyarlılığı %48.00, �zg�ll�ğ� %84.62 olarak bulunurken, MALDI TOF MS'de duyarlılık %80.00, �zg�ll�k %100 olarak bulunmuştur. Bu �alışma sonucunda, VGS'ların tanısının karmaşık bir s�re� olması nedeniyle klinik mikrobiyoloji laboratuvarında bu izolatların tanımlamasında y�ntemlerin tek başına yeterli olamayacağı ve MALDI-TOF MS sisteminin VGS tanısı i�in kullanılabileceğini ancak, gerektiği durumlarda optokin testi ve/veya molek�ler y�ntemlere tanı algoritmasında yer verilmesi gerektiği d�ş�n�lm�şt�r.
Anahtar s�zc�kler: Viridans grups treptokok; MALDI-TOF; 16S rRNA dizi analizi.
ABSTRACT
Accurate identification of viridans group streptococci (VGS) frequently encountered as a causative agent of infective endocarditis is always a challenge for the clinical microbiology laboratory. Clinical microbiology laboratories generally use semi automatic/full automatic systems, molecular methods and also conventional methods for the identification of these bacteria. There are recent published studies that have used MALDI-TOF (Matrix Assisted Laser Ionization Mass Spectrometry-Time of Flight) systems in the identification of VGS. The aim of the study was to compare the performance of the conventional methods, semi automatic and MALDI-TOF MS system used in identification of VGS in oral microbiota of persons under the risk of infective endocarditis, with the gold standard method 16S rRNA sequence analysis and to create a diagnosis algorithm for the identification of VGS in clinical microbiology laboratories according to the obtained data.The study was conducted with 51 VGS� strains isolated from oral microbiota of the patients with rheumatologic cardiac, valve and/or prosthetic valve diseases, under the risk of development of infective endocarditis, who have admitted to Ankara Numune Training and Research Hospital, Department of Cardiology, between February-June 2015. Standard microbiology procedures, optochin susceptibility and bile solubility tests were done for the isolation of bacteria. Bacteria were also identified with APISTREP (bioM�rieux, France) and MALDI-TOF MS Bruker Microflex (Bruker Biotyper; Bruker Daltonics, Bremen, Germany) methods. BSF-8 (5�-AGAGTTTGATCCTGGCTCAG-3�) and BSR-534(5�-ATTACCGCGGCTGCTGGC-3�) primers were used in the 16S rRNA sequence analysis of bacteria. ABI PRISM 3100 Avan t Genetic Analyzer (Applied Biossytems, Foster City, CA, USA) were used for the sequence analysis. Electropherograms were analyzed in SeqScape Software (Applied Biosystems, Foster City, CA, USA) and compared with the reference sequences in GenBank with BLASTN (NCBI). According to the result of optochin and bile solubility tests, with API STREP system, 16 (31,37%) of the isolates were identified as Mitis group, 15 (29.41%) as Anginosus group, 9 (17.5%) as Salivarius group, 7 (13,73%) as Sanguinis group and 4 (7.84%) as Bovis group among optochin and bile resistant alpha hemolytic streptococci. Moreover, of the same isolates 20 (39.22%) were identified as Mitis group, 14 (27.45%) as Anginosus group, 13 (25.49%) as Salivarius group and 4 (7.84%) as Sanguinis group with MALDI-TOF system. In the identification with 16S rRNA, 25 (49.02%) of the isolates were identified as Mitis group, 13 (25.49%) as Anginosus group, 12 (23.53%) as Salivarius group and 1 (1.96%) as Sanguinis group. According to the results, it was determined that 33 (64.70%) of the isolates identified in MALDI-TOF MS system and 31 (60.78%) of the isolates identified in API STREP system were compatible with 16S rRNA sequence analysis method. For Mitis group, API STREP test sensitivity was 48.00% and specificity was 84.62% and MALDI-TOF system sensitivity was 80.00% and specificity was 100%. As VGS identification is a complicated process, we believe a single method will be insufficient for the identification of these isolates in clinical microbiology laboratories. We suggest that MALDI-TOF system can be used for VGS diagnosis, however, optochin test and/or molecular methods should also be included in the diagnosis algorithm when necessary.
Keywords: Viridans group streptococcus; MALDI-TOF; 16S rRNA sequenceanalysis.
Geliş Tarihi (Received): 21.11.2016 - Kabul Ediliş Tarihi (Accepted): 02.01.2017
GİRİŞ
Oral streptokoklar b�y�k oranda viridans grup streptokok (VGS) �yesidirler ve bu bakteriler oral mikrobiyotanın baskın �yelerindendir1. Viridans grup streptokoklar 16S rRNA dizi analizi �alışmalarından elde edilen verilere g�re beş grupta toplanırlar. Gruplar ve bu gruplarda yer alan bazı bakteriler Tablo I'de yer almaktadır2. vMikrobiyota elemanı VGS'ler d�ş�k vir�lans �zelliklerine karşın enfektif endokardit (EE), sepsis, bakteriyemi gibi ciddi enfeksiyonların etkeni olarak karşımıza �ıkabilmektedir. Bu hastaların antibiyotik tedavilerinin planlanabilmesi i�in �rneklerin t�r d�zeyinde tanımlanması �nemlidir3. Bu grup bakterilerin halen taksonomik olarak tanımlanmasında sorunlar yaşanmaktadır4. Etkenin �zellikle Mitis grup i�inde yer alan Streptococcus pneumoniae olup olmadığının belirlenmesi �ok �nemlidir. Optokin ve safrada erime testleri �ok yaygın olarak kullanılan g�venilir testler olmasına karşın testlerin değerlendirilmesi i�in 18-24 saatlik bir s�reye ihtiya� duyulması testlerin en b�y�k dezavantajıdır5.
Son yıllarda, �zellikle ribozomal proteinler başta olmak �zere h�cre proteinlerini baz alarak tanımlama yapan The Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry (MALDI-TOF MS) cihazı mikroorganizmaların tanımlamasında sıklıkla kullanılan g�venilir bir sistemdir6. Sistemin konvansiyonel y�ntemler ile kıyaslandığında hızlı, ucuz ve g�venilir olmak gibi bir�ok �st�nl�ğe sahip olduğu bildirilmekle beraber �zellikle VGS'lerin tanımlanmasında molek�ler y�ntemler ile karşılaştırıldığında sorunlar olduğu belirtilmektedir7. Bu �alışma, EE gelişme riski olan hastaların ağız s�r�nt� �rneklerinden elde edilen optokin ve safraya diren�li oral VGS tanımlanmasında yarı otomatize ve MALDI-TOF MS sistemlerinin 16S rRNA sonu�larına g�re performansını belirlemek ve laboratuvarlarda VGS tanısı i�in bir tanı algoritması oluşturmak ama�lanmıştır.
GERE� ve Y�NTEM
Hastaların Belirlenmesi
Protez kapağı, kapak ve/veya romatizmal kapak hastalıkları olan, Ankara Numune Eğitim ve Araştırma Hastanesi Kardiyoloji Polikliniğine başvuran ve �alışma i�in �rnek vermeyi kabul eden hastalar �alışmaya dahil edildi. �alışma s�recinde son 15 g�n i�inde antibiyotik kullanan, imm�ns�presif ve 18 yaşından k���k hastalar �alışma dışı bırakıldı. Şubat-Haziran 2015 tarihleri arasında �alışmaya dahil edilme kriterlerine uyan 51 hastadan �rnek toplandı. Hastaya zarar vermeden steril ek�vyon ile hastaların diş etleri ve bukkal mukozasından toplanan s�r�nt� �rnekleri ekim yapılana kadar Amies (Coban, Italya) besiyerinde bekletildi.
Bakteri İzolasyonu
Amies besiyerindeki �rnekler oda sıcaklığında bekletilmiş olan %5'lik koyun kanı i�eren agara tek koloni d�şecek şekilde ekildi. Ekimi yapılan plaklar %5 CO2''li ortamda 35 � 1�C'de 18 � 24 saat ink�be edildi. İnk�basyon sonunda %5 koyun kanlı plakta yaygın olarak �remiş olan alfa hemolitik kolonilerden tek koloni pasaj yapıldı. Gram boyamada gram pozitif kok g�r�len kolonilerin taze k�lt�r�nden optokin ve safrada erime testi �alışıldı. Optokin duyarlı ve safrada erime testi pozitif olan izolatlar S.pneumoniae olarak tanımlanarak �alışmaya dahil edilmedi. �alışmaya dahil edilen optokin ve safraya diren�li 51 izolat �alışma g�n�ne kadar -80�C'de skim milk besiyerinde saklandı. T�m sistemlerin kalite kontrol� S.pneumoniae ATCC 49619 suşu ile yapıldı.
API 20 STREP ile Tanımlama
�retici firma �nerileri doğrultusunda bakterilerin API 20 Strep System (bioMerieux, Fransa) kiti ile bakteri tanımlaması yapıldı. 18-24 saatlik ink�basyondan sonra yapılan değerlendirmeler web tabanlı apiWEB sistemi ile değerlendirildi.
MALDI-TOF MS Sistemi ile Tanımlama
Bakterilerin tanımlaması MALDI-TOF MS Bruker Microflex LT model Flex Control 3.0 yazılımı (Bruker Biotyper; Bruker Daltonics, Bremen, Almanya) kullanılarak yapıldı. �rneklerin hazırlanmasında direkt transfer metodu kullanıldı. 24-48 saatlik ink�basyon sonunda tek koloniden k���k bir miktar tahta �ubuk ile plakaya s�r�ld� ve �zerine 1 �l matriks sol�syonu (-cyano-4-hydroxycinnamic acid in 50 acetonitrile/%2.5 trifluoroacetic acid, Bruker Daltonics, Bremen, Almanya) eklendi. Sistemle �alışırken, lineer pozitif iyon modunda 2000-20.000 Da k�tle aralığında, mikroorganizma tanımlama i�in optimize edilmiş uygun y�ntem ile �ift �l��mler ger�ekleştirildi. İyon kaynağı olarak 340 nm'de, 60Hz'lik nitrojen lazer kullanıldı. Spektrumların elde edilmesi i�in her bir numunenin �l��m�nde, toplamda 240 olacak şekilde 40'arlı paketlerden oluşan lazer atışları ger�ekleştirildi. Her bir bakterinin tanımlaması yaklaşık 5 dakikalık bir s�re i�inde tamamlandı.
16S rRNADizi Analizi ile Tanımlama
�alışmaya dahil edilen izolatların t�r d�zeyinde tanımlaması parsiyel 16S rRNA dizi analizi ile yapıldı. �alışmada 16S rRNA'nın 526 baz �iftlik b�lgesi, polimeraz zincir reaksiyonu (PCR) ile �niversal primerler (BSF-8 (5�-AGAGTTTGATCCTGGCTCAG-3�) ve BSR-534 (5�-ATTACCGCGGCTGCTGGC-3�)) kullanılarak amplifiye edildi8. PCR �r�nleri, QIAquick PCR saflaştırma kiti (Qiagen, N.V. Hollanda) ile saflaştırıldı ve "BigDye Direct CycleSequencing Kit (AppliedBiosystems, ABD)" ile amplifiye edildi. Amplikonlar, "ABI PRISM 3100 AvantGenetic Analyzer (AppliedBiosystems,Foster City, CA, ABD)" cihazına y�klenerek dizi analizi ger�ekleştirildi. Elde edilen elektroferogramlar, "SeqScape Software (Applied Biosystems Foster City, CA, ABD)"programı ile analiz edilerek BLASTN (NCBI) programı ile GenBank referans dizileri ile karşılaştırıldı.
İstatistiksel Analiz
Verilerin istatistiksel analizi SPSS 20.0 (Inc. ABD) programı kullanılarak yapıldı. Testlerin duyarlılık ve �zg�ll�ğ� McNemar testi kullanılarak belirlendi.
BULGULAR
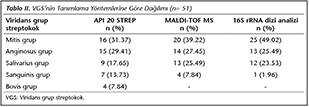
API 20 STREP yarı otomatize sistemden elde edilen sonu�lar apiweb sistemine y�klenerek %70'in �zerinde uyumlu olan sonu�lar alınmıştır. MALDI-TOF MS sistemi ile yapılan değerlendirmelerde > 1.7 skoru baz alınmış ve bu skorun �zerindeki tanımlamalar doğru olarak kabul edilmiştir.16S rRNA dizi analizi ile yapılan değerlendirmede GenBank'ta yayımlanmış dizi analizlerine g�re ≥ %99 ve ≥ %96 oranında gen dizi benzerliği bulunan 51 VGS izolat t�r d�zeyinde tanımlanmıştır. Bu değerlendirmelere g�re, t�r d�zeyinde tanımlanmış olan VGS'lar gruplara ayrılmıştır ve testlerin performans değerlendirmeleri gruplar �zerinden yapılmıştır. Grupların tanı sistemlerine g�re dağılımı Tablo II'de yer almaktadır. MALDI-TOF MS sistemi ile Mitis grup i�indeki 2 (%10.0) izolat S.pneumoniae olarak tanımlanmıştır. Buna karşın, 16S rRNA dizi analizi ile yapılan �alışmada hi�bir izolat S.pneumoniae olarak tanımlanmamıştır. MALDI-TOF MS sistemi ve API 20 STREP Sistemleri ile yapılan �alışmalardan elde edilen sonu�ların 16S rRNA tanımlanmasına g�re duyarlılık, �zg�ll�k, pozitif prediktif ve negatif prediktif değerleri Tablo III'te yer almaktadır.
TARTIŞMA
Literat�rde VGS tanımlanmasına y�nelik test performanslarının değerlendirildiği bir�ok benzer �alışma yer almaktadır. Viridans streptokok grubunun en �nemli �yesi tartışmasız S.pneumoniae olup �alışmalar genellikle bu bakteri �zerinde yapılmıştır. �alışmamızda �zellikle konvansiyonel y�ntemler ile S.pneumoniae olarak tanımlanmış olan izolatlar dışarıda bırakılarak test y�ntemlerinin daha �ok diğer VGS'nin tanımlanmasındaki performansını değerlendirmek ama�lanmıştır.
�lkemizdeki klinik mikrobiyoloji laboratuvarlarında bakteri tanımlamasında yarı otomatize sistemler yaygın olarak kullanılmaktadır. Her laboratuvarın ileri teknikleri kullanma olasılığı yoktur ve ayrıca her laboratuvarın otomatize ve molek�ler y�ntemleri kullanması maliyet etkin değildir. Bu �alışmada, 16S rRNA gen dizi analizi ile VGS bakterilerinin tanımlamasında kullanılan testlerin performansını değerlendirmeyi ve bu sistemleri kullanan laboratuvarlarda VGS tanımlamasında nasıl bir yol izlemeleri gerektiğini ortaya koymaya �alıştık.
Mitis grup, VGS'ler arasında klinik �rneklerden en sık izole edilen grup olarak karşımıza �ıkmaktadır9,10. �alışmamızda Mitis grubunun florada en yaygın olan VGS grubu olduğunu belirlenmiştir. Kan k�lt�r�nde etken olarak izole edilen VGS'lerin tanımlamasında API 20 STREP sisteminin kullanımının g�venilir sınırlarda olmadığı s�ylenebilir11,12. Elde edilen verilere g�re, VGS tanımlamasında yarı otomatize sistem kullanan laboratuvarların raporlarını bir �n rapor olarak hazırlamaları ve izolatları bir referans laboratuvara g�ndererek doğrulama yapılması daha doğru bir yaklaşım olacaktır.
Klinik mikrobiyoloji laboratuvarlarında hızlı tanı testlerinin yeri g�n ge�tik�e artmaktadır. ��nk� enfeksiyonlu bir hastanın tedavisinin doğru y�netilmesi i�in hız bir zorunluluk olarak karşımıza �ıkmaktadır. Bu ama�la, MALDI-TOF MS sistemleri klinik mikrobiyoloji laboratuvarlarında kullanıma girmiştir. Hızlı tanı testi olması ve bunun bir sonucu olarak mortalite ve hasta yatış s�resinde azalmaya katkıda bulunduğu ka�ınılmaz bir ger�ektir13. Bu �alışmada, MALDI-TOF MS sisteminin �zellikle Mitis grubu ve Anginosus grubu tanısı i�in kabul edilebilir g�ven aralığında yer aldığı s�ylenebilir. �zellikle bu iki grubun VGS enfeksiyonlarında g�r�len en sık etkenler olması bu y�ntemin kabul edilebilirliğini de arttırabilir.
Bu �alışmada, optokin duyarlı ve safrada erime testleri ile tanımlanan S.pneumoniae izolatları �alışma dışında tutulmuştur. BrukerBiotyper MS sisteminde S.pneumoniae izolatı i�in skorun > 2 olması durumunda bile izolatın mutlaka ya optokin duyarlılığının belirlenmesi ya da dizi analizi ile doğrulanması gerekmektedir14. 16S rRNA dizi analizi �alışmalarında S.pneumoniae, S.pseudopneumoniae, S.mitis ve S.oralis ile �ok y�ksek benzerlikler g�stermekte olup, t�r d�zeyinde tanımlama olduk�a zor olmaktadır15,16. Son zamanlarda yapılmış olan �alışmalar,S.pneumoniae'nın 16S rRNA gen dizisinde 203. pozisyonda bir sitozin i�erdiğini, diğer Mitis grubu �yelerinde bu pozisyonda adeninin yer aldığı bildirilmektedir14,17
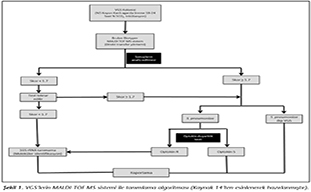
VGS'lerin t�r d�zeyinde doğru tanımlanması patojenite ve vir�lans fakt�rlerinin belirlenmesi i�in de �nemlidir.� Bu durum, �zellikle S.pneumoniae'nın i�inde yer aldığı Mitis grubunun tanımlanmasında daha b�y�k bir �nem taşımaktadır. BrukerBiotyper MS sisteminde pn�mokok olmayan izolatların sıklıkla S.pneumoniae olarak tanımlandığı ve bu sistemin �zellikle bu grup i�in performansının d�ş�k olduğu bildirilmektedir11,18. �alışmamızda optokin duyarlı pn�mokok izolatları �alışmanın başında dahil edilmediği i�in sistemin performansının Mitis grup i�in bu nedenle �ok d�ş�k bulunmadığını d�ş�n�yoruz. Doğrudan transfer y�ntemi ile S.pneumoniae, S.mitis ve S.oralis tanımlamasının yapıldığı bir �alışmada,tanı olasılığını arttırmak i�in referans suşlar ile bir k�tle spektrum modeli geliştirilmiş ve doğrudan transfer y�nteminde doğru tanımlama oranının %66.7'den, %100'e y�kseldiği saptanmıştır19. Bilindiği �zere, dizi analiz y�ntemleri hem �ok pahalı hem de zaman alıcı y�ntemlerdir. Optokin testi ise ucuz ve rutin laboratuvarlarda rahatlıkla kullanılabilecek bir test olup �ok nadiren yanlış bakteri tanımlamasına neden olmaktadır20. VGS i�in hızlı tanı testlerinin g�venirliliğini arttırmaya y�nelik tanı algoritmalarının hazırlanmasının ya da spektrum modeli geliştirilmesinin faydalı olacağı kanısındayız. Klinik laboratuvarda VGS'nin tanımlanması amacıyla elde edilen veriler doğrultusunda başka bir �alışma sonucunda oluşturulan tanı algoritmasının bizim verilerimizle de uygun olduğu bulunmuştur14. �alışmadan ve elde edilen verilerden esinlenerek hazırlanan tanı algoritması Şekil 1'de yer almaktadır. Algoritmaya g�re MALDI-TOF MS sisteminde tanı skoru� ≥ 1.7 olan tanımlamaların S.pneumoniae hari� raporlanabileceği, ancak S.pneumoniae i�in skorun > 2 olması durumunda bile, mutlaka optokin duyarlılığının g�sterilmesi gerektiği belirtilmiştir. Skoru < 1.7 olan tanımlamalar i�in aynı y�ntem ile testin tekrar edilmesi ve skoru > 2 olan optokine diren�li bulunan S.pneumoniae izolatları i�in ise 16S rRNA dizi analizi sonucuna g�re raporlandırmanın yapılması gerektiği kanısındayız.
VGS tanısı karmaşık bir s�re� olup, klinik mikrobiyoloji laboratuvarında bu izolatların tanısında y�ntemlerin tek başına kullanımının yeterli olamayacağını d�ş�n�yoruz. MALDI-TOF MS ve API STREP sistemleri kullanıldığında konvansiyonel y�ntemler ve/veya molek�ler y�ntemlere de tanı algoritmasında yer verilmeli ve gerektiğinde referans laboratuvarlarda izolatların doğru tanımlanması ger�ekleştirilmelidir.
KAYNAKLAR
- Whiley RA, Fraser HY, Douglas CWI, et al. Streptococcus parasanguis sp. Nov.: An atypical viridans Streptococcus from human clinical specimens. FEMS Microbiol Lett 1990; 56(1-2): 115-21.
- Ergin A. Viridans streptokokların laboratuvar tanısında klasik ve yeni yaklaşımlar. Mikrobiyol Bul 2010; 44(3): 495-503.
- Doern CD, Burnham CA. It's not easy being green: the viridans group streptococci, with a focus on pediatric clinical manifestations. J Clin Microbiol 2010; 48(11): 3829-35.
- Teles C, Smith A, Ramage G, Lang S. Identification of clinical lyrele vantviridans group streptococci byphenotypic and genotypicanalysis. Eur J Clin Microbiol Infect Dis 2011; 30(2): 243-50.
- Kellogg JA, Bankert DA, Elder Carol J, Gibbs J, Smith MC. Identification of Streptococcus pneumoniae revisited. J Clin Microbiol 2001; 39: 3373-5.
- Neville SA, Lecordier A, Ziochos H, et al. Utility of matrix-assisted laser desorptionion ization-time of flight mass spectrometry following introduction for routine laboratory bacterial identification. J Clin Microbiol 2011; 49(8): 2980-4.
- Sasaki E, Osawa R, Nishitani Y, Whiley RA. Development of a diagnostic PCR assay targeting themn dependent superoxide dismutase gene (sodA) for identification of Streptococcus gallolyticus. J Clin Microbiol 2004; 42(3): 1360-2.
- Kemp M, Bangsborg J, Kjerulf A, et al. Advantages and limitations of ribosomal RNA PCR and DNA sequencing for identification of bacteria in cardiac valves of danish patients. Open Microbiol J 2013; 7: 146-51.
- Ikryannikova LN, Filimonova AV, Malakhova MV, et al. Discrimination between Streptococcus pneumoniae and Streptococcus mitis based on sorting of their MALDI mass spectra. Clin Microbiol Infect 2013; 19(11): 1066-71.
- Angeletti S, Dicuonzo G, Avola A, et al. Viridans Group Streptococci clinical isolates: MALDI-TOF mass spectrometry versus gene sequence-based identification. PLoS One 2015; 10(3): e0120502-12.
- L�pez Roa P, S�nchez Carrillo C, Mar�n M, Romero F, Cercenado E, Bouza E. Value of matrix-assisted laser desorption ionization-time of flight for routine identification of viridans group streptococci causing bloods tream infections. Clin Microbiol Infect 2013; 19(5): 438-44.
- Chang YC, Lo HH. Identification, clinical aspects, susceptibility pattern, and molecular epidemiology of beta-haemolytic group G Streptococcus anginosus group isolates from central Taiwan. Diagn Microbiol Infect Dis 2013; 76(3): 262-5.
- Angeletti S. Matrixassisted laser desorption time of flight mass spectrometry (MALDI-TOF MS) in clinical microbiology. J Microbiol Methods 2016; 7012(16): 30253-6.
- Zhou�M, Yang� Q, Kudinha T, et al. Using Matrix-Assisted Laser Desorption Ionization-Time of Flight (MALDI-TOF) Complemented with Selected 16S rRNA and gyrB Genes Sequencing to Practically Identify Clinical Important Viridans Group Streptococci (VGS). Front Microbiol 2016; 7: 1328-35.
- Suzuki N, Seki M, Nakano Y, Kiyoura Y, Maeno M, Yamashita Y. Discrimination of Streptococcus pneumoniae from viridans group streptococci by genomic subtractive hybridization. J Clin Microbiol 2005; 43(9): 4528-34.
- Haanper� M, Jalava J, Huovinen P, Meurman O, Rantakokko-Jalava K. Identification of alpha-hemolytic streptococci by pyrosequencing the 16S rRNA gene and by use of VITEK 2. J Clin Microbiol 2007; 45(3): 762-70.
- Scholz�CF,� Poulsen� K,� Kilian� M. Novel molecular method for identification� of� Streptococcus pneumoniae applicable to clinical microbiology. and 16S rRNA sequence-based microbiome studies. J Clin Microbiol 2012; 50(6): 1968-73.
- Ikryannikova LN, Lapin KN, Malakhova MV, et al. Misidentification of alpha-hemolytic streptococci by routine tests in clinical practice. Infect Genet Evol 2011; 11(7): 1709-15.
- Chen JH, She KK, Wong OY, et al. Use of MALDI Biotyperplus Clin Pro Tools mass spectra analysis for correct identification of Streptococcus pneumoniae and Streptococcus mitis/oralis. J Clin Pathol 2015; 68(8): 652-6.
- Ko KS, Oh WS, Peck KR, Lee JH, Lee NY, Song JH. Phenotypic and genotypic discrepancy of Streptococcus pneumoniae strains isolated from Asian countries. FEMS Immunol Med Microbiol 2005; 45(1): 63-70.
İletişim (Correspondence):
Uzm. Dr. Serap S�z�k Yıldız,
T�rkiye Halk Sağlığı Kurumu,
Mikrobiyoloji Referans Laboratuvarları Daire Başkanlığı,
Adnan Saygun Caddesi No: 55,
06100 Sıhhiye, Ankara, T�rkiye.
Tel (Phone): +90 312 565 5771,
E-posta (E-mail): serapsuzuk@gmail.com